| Autore |
Discussione |
|
|
Schuldiner86
Utente Junior

Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 29 novembre 2008 : 18:54:54 Inserito il - 29 novembre 2008 : 18:54:54






|
Ragazzuoli, sono disperato...Ho passato il pomeriggio a cercare la sequenza del vettore dell'Invitrogen pCR II-TOPO, ma ho trovato solo la variante Blunt che non è quello che mi serve...Sapreste indicarmi un database che contiene le sequenze di tutti i vettori, o dal sito dell'invitrogen dove posso reperire certe informazioni?
La sequenza mi serve per disegnare una mappa decente del vettore che mi serve per una illustrazione della tesi...A tal proposito mi sapreste indicare per favore un programma che disegni delle mappe belle esteticamente? Ho trovato pDRAW ma non mi piace moltissimo...
Altrimenti mi sapreste spiegare come potrei fare a disegnarne uno con Photoshop con tutti gli effetti? Grazie mille in anticipo a chiunque vorrà aiutarmi...
|
 
 |
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 29 novembre 2008 : 19:54:04 Inserito il - 29 novembre 2008 : 19:54:04




|
| Per i software specifici non ti so dire, ma direi di lasciar perdere PS per queste cose, usa un programma di grafica vettoriale, suggerisco caldamente Inkscape |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 novembre 2008 : 20:25:40 Inserito il - 29 novembre 2008 : 20:25:40





|
Citazione:
Messaggio inserito da domi84
...da CLC Sequence Viewer (di cui parlavo qui: http://mybioinformatica.wordpress.com/2008/10/13/editor-di-sequenze-clc-sequence-viewer/ ) e vedi la sequenza nella forma circolare...questo è il risultato (vedi sotto)
Oooooo  , ne parlavi sul blog!!! , ne parlavi sul blog!!!
Non sapevo di questa funzionalità di CLC Sequence Viewer! Dovrei seguirlo di più il tuo blog Dovrei seguirlo di più il tuo blog  ... mi sa che sono rimasta alla lezione 2 o 3! ... mi sa che sono rimasta alla lezione 2 o 3! 
Citazione:
P.S. Forse queste cose dovrei scriverle sul blog...
Si decisamente!!! |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 29 novembre 2008 : 21:04:32 Inserito il - 29 novembre 2008 : 21:04:32






|
| Vi ringrazio infinatamente a tutti...Non so perché sono stato così stupido da non trovare la sequenza sul sito dell'invitrogen, forse ero troppo disperato... |
 
 |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 29 novembre 2008 : 21:23:49 Inserito il - 29 novembre 2008 : 21:23:49






|
| CLC mi sembra veramente potente...una cosa sola...Se volessi mettere le frecce per i marker e gli ORF e i rettangoli per le origini con le annotazioni come devo fare? |
 
 |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 30 novembre 2008 : 01:50:47 Inserito il - 30 novembre 2008 : 01:50:47




|
Purtroppo indicavo proprio questo come difetto di CLC. Non essendo un programma libero, la versione in grado di modificare le annotazioni è a pagamento . .
Nel tuo caso potresti scaricare la versione a pagamento e provarla per 30 giorni, quindi fare le tue modifiche.
Altrimenti puoi modificare il file di testo ottenuto da Genbank.
Oppure ancora puoi prendere l'immagine ottenuta da CLC e modificarla con Inkscape. |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 30 novembre 2008 : 14:52:18 Inserito il - 30 novembre 2008 : 14:52:18






|
Peccato perché mi piaceva proprio come venivano... Sono riuscito a fare qualcosa con pDraw ma mi sembra tanto da bambino, e poi volevo una cosa tipo con gli effetti di luce sulle frecce degli ORF...Posto l'immagine...Secondo coi è una bambinata? Sono riuscito a fare qualcosa con pDraw ma mi sembra tanto da bambino, e poi volevo una cosa tipo con gli effetti di luce sulle frecce degli ORF...Posto l'immagine...Secondo coi è una bambinata? 
 |
 
 |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 30 novembre 2008 : 17:13:22 Inserito il - 30 novembre 2008 : 17:13:22




|
Citazione:
Secondo coi è una bambinata?
mio nipote disegna plasmidi tutti i giorni!  
No, a parte gli scherzi, ti do un paio di suggerimenti
1) usa bordi più fini
2) non usare bordi neri. Usa un tono più scuro del colore della freccia
3) se vuoi usa un piccolo gradiente
4) usa colori pastello invece di colori "forti" (ok, qui forse si va su gusti personali)
Comunque ho fatto una prova veloce con inkscape, direi che così è più carino no?
Immagine:
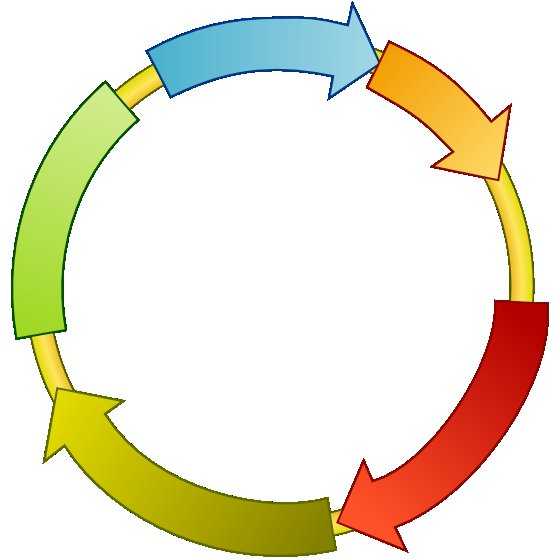
30,6 KB
Allegato:  plasmid.svg.zip plasmid.svg.zip
85,9 KB |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 30 novembre 2008 : 17:18:20 Inserito il - 30 novembre 2008 : 17:18:20






|
| Si si, è proprio di gran lunga meglio...Mi dedicherò a InkScape questi giorni...E' pDraw che usa quei bordi, il tuo a confronto è un'opera d'arte...Grazie mille a tutti per tutte le dritte! |
 
 |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 30 novembre 2008 : 18:11:11 Inserito il - 30 novembre 2008 : 18:11:11






|
| Thanks...Anche perché ci sto smanettando adesso per la prima volta, ma oltre a disegnare un cerchio non riesco andare oltre...Penso proprio di aver bisogno di un manuale...La grafica non ha mai fatto per me!!! =D |
 
 |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Città: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 30 novembre 2008 : 18:21:14 Inserito il - 30 novembre 2008 : 18:21:14





|
Perche' non provate anche questo sito:
- http://www.biorubyrails.com/openlab
Dalle immagini sembra essere in grado di fare grafici come quelli che richiedi.
 |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 30 novembre 2008 : 18:33:39 Inserito il - 30 novembre 2008 : 18:33:39






|
| Sul sito sembra ci siano soltanto mappe già belle e pronte e non strumenti per crearle... |
 
 |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Città: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 30 novembre 2008 : 18:48:46 Inserito il - 30 novembre 2008 : 18:48:46





|
E' la prima volta che lo uso, e non sono esperto di cose di laboratorio :)
Pero' mi sembra che tu debba crearti un account (gratuito) e dopo loggarti; da li' vai su Data Center > Sequences e clicchi su 'nuova sequenza', indicandola come circolare;
infine ci clicchi sopra, e ti ritrovi un menu' in cui puoi aggiungere manualmente i marker, indicando posizione di inizio e fine.
 |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Città: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 30 novembre 2008 : 19:00:55 Inserito il - 30 novembre 2008 : 19:00:55





|
Anzi, secondo me questo e' il sistema piu' veloce per fare quello che hai chiesto.
I consigli che ti hanno dato sono molto utili (a me da ignorante, sono piaciuti molto quelli di Chick) pero' se hai tanta fretta, con questo fai in un attimo.
Non hai nemmeno bisogno di avere la sequenza del plasmido, puoi crearne una con 3793 N o basi casuali. |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 30 novembre 2008 : 19:15:19 Inserito il - 30 novembre 2008 : 19:15:19




|
Certo, chiaramente se hai qualcosa che fa già il tutto è più veloce. Inkscape o programmi simili hanno il vantaggio di darti un'estetica migliore.
Per facilitarti il tutto puoi creare l'immagine con un software fatto a posta per disegnare plasmidi, importare l'immagine in inkscape e usarla come base di riferimento.
Se uno poi avesse tanta voglia... inkscape supporta plugins scritti in python e si potrebbe fare una cosa che crea la mappa in automatico... quindi maghi del python mettetevi all'opera!
Se vuoi dei tutorial per inkscape suggerisco di guardare qui:
http://screencasters.heathenx.org/ |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Città: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 30 novembre 2008 : 19:26:44 Inserito il - 30 novembre 2008 : 19:26:44





|
Citazione:
Se uno poi avesse tanta voglia... inkscape supporta plugins scritti in python e si potrebbe fare una cosa che crea la mappa in automatico... quindi maghi del python mettetevi all'opera!
mmmm... se mi dici esattamente cosa si deve fare, ci posso dare una occhiata... |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 30 novembre 2008 : 20:16:08 Inserito il - 30 novembre 2008 : 20:16:08






|
dallolio scusa ma sto svalvolando io...Non lo so perché ma alle cose stupide ultimamente non ci arrivo più, come arrivare a loggarmi...Comunque mi sono scaricato qualche tutorial di InkScape e sembra davvero carino come programma, soprattutto mi piacciono i vari effetti che si possono dare...Appena posso ci do una buona occhiata, altrimenti userò qualche sito che lo fa in automatico, ne avevo trovati anche altri...
EDIT: Altra alternativa interessante...Ci stanno siti come PlasMapper o Savvy che permettono di costruire mappe di plasmidi e come output danno un file .SVG che può essere modificato a piacimento con InkScape... |
 
 |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 02 dicembre 2008 : 11:46:49 Inserito il - 02 dicembre 2008 : 11:46:49






|
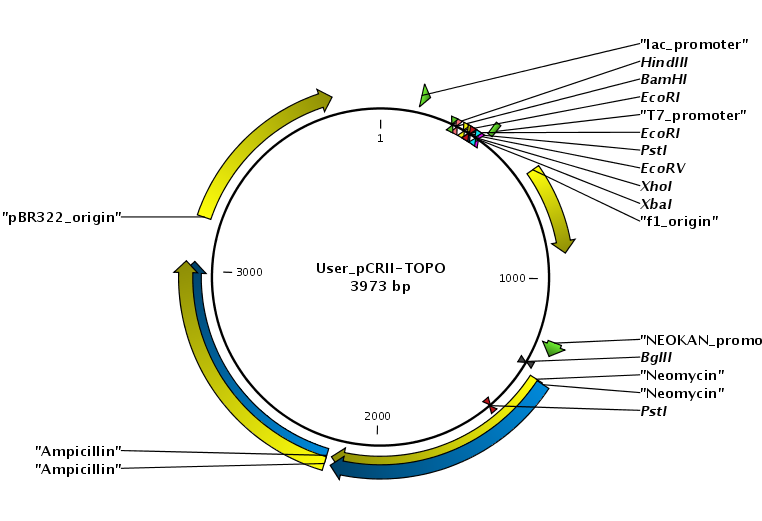
Alla fine ce l'ho fatta con l'ultimo metodo...Jo disegnato il plasmide con Savage Plasmids che dà come output un immagine .svg facilmente modificabile con InkScape...Il risultato è questo...
 |
 
 |
 |
|
|
Schuldiner86
Utente Junior


Prov.: Viterbo
Città: Bologna

581 Messaggi |
 Inserito il - 08 dicembre 2008 : 20:13:35 Inserito il - 08 dicembre 2008 : 20:13:35






|
Altro problemino...Non riesco a trovare la sequenza né la mappa del plasmide pEF-BOS.puro, contenente cioè la resistenza alla puromicina...Potreste darmi una mano per favore? |
 
 |
 |
|
| |
Discussione |
|

















 Python
Python
