| Autore |
Discussione |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 31 maggio 2009 : 22:50:16 Inserito il - 31 maggio 2009 : 22:50:16




|
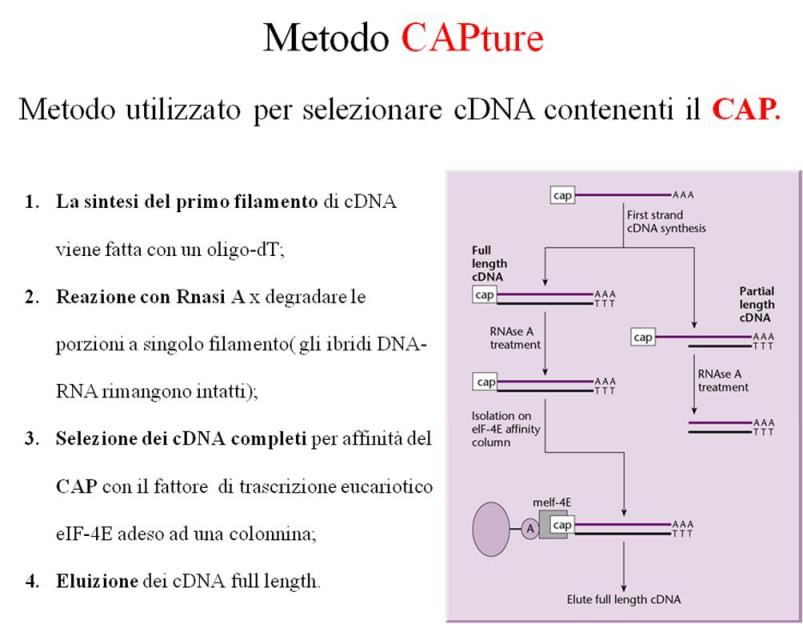
Salve ragazzi,
qualcuno dei genetisti potrebbe spiegarmi perchè nell'oligoCapping per la sintesi di cDNA full-lenght, anche se ci dovesse essere un mispriming (appaiamento dell'oligodT, adoperato per la sintesi del primo filamento di cDNA),non si avrebbe amplificazione?
Perchè come dice l'immagine solo le molecole contenenti ambo i primers verrebbero amplificate?
Vi allego una immagine sperando che qualcuno di buona volontà possa rispondermi.
Grazie
Immagine:
53,98 KB
Immagine:

81,42 KB
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 01 giugno 2009 : 11:50:23 Inserito il - 01 giugno 2009 : 11:50:23





|
Ciao!
Ho ridotto le immagini perché erano un po' enormi.
Citazione:
Messaggio inserito da tambua
... perchè nell'oligoCapping per la sintesi di cDNA full-lenght, anche se ci dovesse essere un mispriming (appaiamento dell'oligodT, adoperato per la sintesi del primo filamento di cDNA),non si avrebbe amplificazione?
Perchè come dice l'immagine solo le molecole contenenti ambo i primers verrebbero amplificate?
Perché durante la reazione di retrotrascrizione può avvenire che l'oligodT si appai in una regione sbagliata, ricca di A (mispriming) e la retrotrascrittasi non ha problemi ha sintetizzare il filamento opposto è molto più permissiva della Taq. Invece durante la PCR è più difficile ottenere una amplificazione se l'appaiamento non è corretto
|
 |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 01 giugno 2009 : 13:52:29 Inserito il - 01 giugno 2009 : 13:52:29




|
Grazie GFPina (che nick fantastico!),
ma ciò che non mi è chiaro è:
"Ma i cDna derivanti da mispriming non verranno amplificati comunque dal primer che si annila alla regione dell'oligo-Cap?
Mi auguro di non scocciarti, ma la cosa come avrai capito mi sta prendendo molto. |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 01 giugno 2009 : 14:59:18 Inserito il - 01 giugno 2009 : 14:59:18





|
Citazione:
Messaggio inserito da tambua
Grazie GFPina (che nick fantastico!),
Grazie! 
Citazione:
Messaggio inserito da tambua
ma ciò che non mi è chiaro è:
"Ma i cDna derivanti da mispriming non verranno amplificati comunque dal primer che si annila alla regione dell'oligo-Cap?
Per avere amplificazione devono essere sintetizzati entrambi i filamenti! Se anche il primer si attacca all'oligo-CAP non c'è il primer reverse che si appaia sul filamento complementare, si formerebbe solo un filamento! |
 |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 02 giugno 2009 : 09:53:03 Inserito il - 02 giugno 2009 : 09:53:03




|
Immagine:

64,97 KB
Ti mando un disegno di ciò che la mia mente bacata aveva ipotizzato, e se possibile dimmi dove sta l'errore.
Ciao GFPina |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 03 giugno 2009 : 10:53:25 Inserito il - 03 giugno 2009 : 10:53:25





|
 Ehm... come l'hai disegnata tu sembra funzionare... Ehm... come l'hai disegnata tu sembra funzionare...
ho provato a cercare un po' ma in effetti da nessuna parte è spiegata questa cosa dicono solo (come dicevo io):
Citazione:
Only full-length cDNAs annealing to both primers will be amplified, thus eliminating broken or degraded RNAs, incomplete fast cDNA strands (which lack a 5' primer annealing site) and misprimed cDNAs (which lack a 3' primer annealing site).
l'articolo originale è questo: Oligo-capping: a simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides.
non so se li sia spiegato, non ho accesso, la mia università ha accesso a questa rivista dall'anno successivo!  |
 |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 03 giugno 2009 : 16:56:20 Inserito il - 03 giugno 2009 : 16:56:20




|
Grazie GFpinuccia proverò a procurarmi l'articolo, altrimenti porrò la domanda al Prof. (ovviamente prima dell'esame e non durante  ). ).
Se o io o tu dovessimo trovare la soluzione al quesito la metteremo qua, magari sarà d'aiuto a chi l'oligocapping non solo lo studia ma lo pratica.
|
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 03 giugno 2009 : 20:37:00 Inserito il - 03 giugno 2009 : 20:37:00





|
Si direi che la cosa migliore è chiedere al Prof!
Aspetto notizie! 
 |
 |
|
| |
Discussione |
|








