Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 06 novembre 2009 : 23:11:41 Inserito il - 06 novembre 2009 : 23:11:41




|
buona sera a tutti.
Mi trovo a fare uno studio di gene expression, più precisamente: mi interessa sapere se un farmaco cambia l'espressione di un gene.
Vi riporto l’esperimento
C1. Controllo non trattato alle 48 h
C2. Trattamento con farmaco 100 µg/µl 48 h
C3. Trattamento con farmaco 200 µg/µl 48 h
C4. Trattamento con farmaco 1000 µg/µl 48 h
C5. Controllo non trattato alle 72 h
C6. Trattamento con farmaco 100 µg/µl 72 h
C7. Trattamento con farmaco 200 µg/µl 72 h
C8. Trattamento con farmaco 1000 µg/µl 72 h
Ho effettuato una qualitativa ed in effetti, trattando le mie cellule con il farmaco di riferimento, osservo una modulazione nell’espressione del gene d’interesse, alle varie [].
Vorrei effettuare una Real time per quantificare l’espressione nelle varie condizioni.
Non ho assolutamente esperienza di questa tecnica.
Come reagente ho a disposizione il Sybr Green
Come devo impostare l’esperimento?
Come riferimento devo utilizzare un gene di controllo? Io ho a disposizione la #946;-actina.
Ma come faccio a controllare l'andamento dei trattamenti rispetto ai controlli non trattati.
Ed il bianco?
Devo lavorare, necessariamente in doppio?
La real time è uno step one della Applied.
Io avrei pensato a questa disposizione:
Immagine:
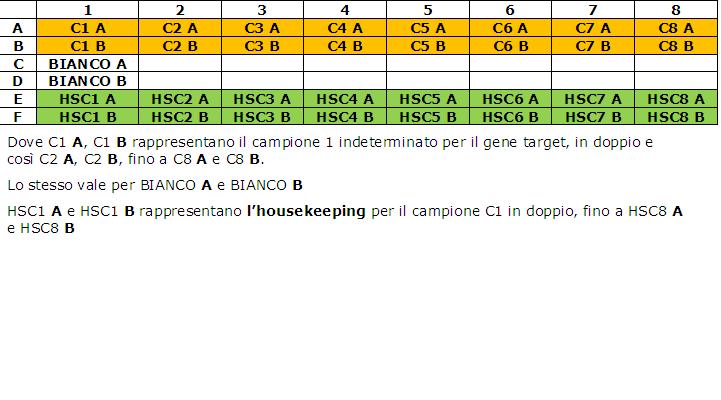
51,09 KB
aiutatemi. mi sto sbattendo la testa su libri e manuali ma non ho alcuna certezza.
grazie.
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 07 novembre 2009 : 00:37:48 Inserito il - 07 novembre 2009 : 00:37:48





|
Beh sulla Real-Time ci sono moltissime discussioni sul forum, hai provato a cercare un po'?
Io personalmente ho scritto trattati, sopratutto sull'utilizzo degli housekeeping e non vorrei ripetermi, ma trovi tutto cercando.
Rispondo solo brevemente alle tue domande:
Citazione:
Come reagente ho a disposizione il Sybr Green
Come devo impostare l’esperimento?
il Sybr Green va bene, dovresti avere una mix che contiene tutto (in genere è così)
devi disegnarti dei primers per real time, o puoi provare ad utilizzare quelli che hai usato per la quantitativa e testarli per assicurati che non diano prodotti aspecifici o dimeri di primers che interferiscano con la lettura, questo lo vedi analizzando la curva di melting.
Citazione:
Come riferimento devo utilizzare un gene di controllo? Io ho a disposizione la #946;-actina.
si devi utilizzare un gene di riferimento che sia espresso allo stesso livello in tutti i campioni, gene housekeeping e poi fare una quantificazione relativa.
Beta-actina è molto utilizzata, ma devi assicurarti che sia veramente housekeeping in tutti i tuoi campioni. (ci sono varie discussioni in proposito)
Citazione:
Ed il bianco?
Il bianco ti serve per vedere che non ci sia fluorescenza aspecifica, essenzialmente fai la stessa cosa che fai per il bianco in una PCR end-point, poi dovresti avere una curva che rimane a livello del background e anche analizzando la curva di melting non dovresti avere alcun picco, altrimenti c'è qualcosa che non va, ad es, dimeri di primers. Poi alcuni programmi prevedono l'utilizzo del bianco e altri no.
Citazione:
Devo lavorare, necessariamente in doppio?
si assolutamente! Anzi sarebbe meglio in triplicato!
Citazione:
La real time è uno step one della Applied.
mmm... vuoi dire che fai una RT-PCR one-step, cioè retrotrascrizione e PCR in un unica provetta?
Io non la amo particolarmente, ma puoi farlo.
Citazione:
Io avrei pensato a questa disposizione:
beh per la disposizione vedi tu, falla come sei più comodo!
 |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 08 novembre 2009 : 08:15:01 Inserito il - 08 novembre 2009 : 08:15:01




|
grazie. 
Citazione:
il Sybr Green va bene, dovresti avere una mix che contiene tutto (in genere è così)
devi disegnarti dei primers per real time, o puoi provare ad utilizzare quelli che hai usato per la quantitativa e testarli per assicurati che non diano prodotti aspecifici o dimeri di primers che interferiscano con la lettura, questo lo vedi analizzando la curva di melting.
si nel sybr green c'è tutto. c'è anche il ROX. Ma cosa serve?
Relativamente ai primer utilizzerò quelli utilizzati per la PCR qualitativa. che differenza c'è tra questi e primer per real time?
come si analizza la curva di melting?
Citazione:
Beta-actina è molto utilizzata, ma devi assicurarti che sia veramente housekeeping in tutti i tuoi campioni. (ci sono varie discussioni in proposito)
cosa ne pensi a proposito di un RNA mitocondriale come hk?
Citazione:
mmm... vuoi dire che fai una RT-PCR one-step, cioè retrotrascrizione e PCR in un unica provetta?
Io non la amo particolarmente, ma puoi farlo.
Non è una one step PCR. Lo strumento che utilizziamo si chiama SPEPONE, della Applied.
Grazie, veramente di tutti i consigli.
Mi piace perchè, con questo forum, si ha la sensazione di lavorare in un laboratorio comune, sicuramente virtuale, ma dove le esperienze di ciascuno vengono messe a disposizione di tutti.
Io, per quanto posso essere utile, metto a disposizione la mia breve esperienza nel campo della clinica ematologica ed oncologica.
a presto  |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 08 novembre 2009 : 09:51:53 Inserito il - 08 novembre 2009 : 09:51:53




|
Citazione:
si nel SYBR Green c'è tutto. c'è anche il ROX. Ma cosa serve?
E' un colorante di riferimento: ti serve, prima di iniziare l'amplificazione, per normalizzare tutte le provette secondo la fluorescenza del ROX (visto che non avrai sufficiente fluorescenza dal SYBR Green) |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 08 novembre 2009 : 15:43:30 Inserito il - 08 novembre 2009 : 15:43:30





|
Per il ROX ti ha già spiegato Nico, ti faccio solo presente giusto per conoscenza che non tutti i macchinari richiedono l'utilizzo di ROX, ma in genere le mix sono formulate apposta per il macchinario sul quale le devi utilizzare.
Piccola nota:
Citazione:
si nel sybr green c'è tutto. c'è anche il ROX.
Nella "Mix" c'è tutto oltre al SYBR Green!
Il SYBR Green di per sé è un colorante fluorescente che puoi comprarti anche a parte (in teoria potresti utilizzare una mix di PCR normale e aggiungerci SYBR Green)
Citazione:
cosa ne pensi a proposito di un RNA mitocondriale come hk?
Non saprei RNA mitocondriale non l'ho mai visto utilizzare come housekeeping! Tra i più stabili ci sono gli RNA ribosomiali o che codificano per proteine ribosomiali, io ad es. utilizzo RPL13.
Citazione:
Lo strumento che utilizziamo si chiama SPEPONE, della Applied
ah ok! Ho controllato è StepOne. Beh come strumento non lo conosco, ma quelli di Applied dovrebbero essere buoni.
Citazione:
Relativamente ai primer utilizzerò quelli utilizzati per la PCR qualitativa. che differenza c'è tra questi e primer per real time?
Beh come primer in sé niente, solo devi avere un po' più cautela. In genere è consigliato che amplifichino un frammento non troppo grande, io in genere sto attorno alle 200bp. E se usi SYBR Green è fondamentale che non formino dimeri altrimenti la fluorescenza emessa dai dimeri di primer ti si somma a quella del tuo amplificato e ti si "falsificano" tutti i dati.
Appunto per questo devi analizzare la curva di melting.
Citazione:
come si analizza la curva di melting?
vedi le varie discussioni in proposito, ad es. questa: http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=2538
 |
 |
|
|
farmacologia
Nuovo Arrivato


100 Messaggi |
 Inserito il - 09 novembre 2009 : 07:05:58 Inserito il - 09 novembre 2009 : 07:05:58




|
come al solito grazie   |
 |
|
|
angemore
Nuovo Arrivato

82 Messaggi |
 Inserito il - 09 novembre 2009 : 19:29:14 Inserito il - 09 novembre 2009 : 19:29:14




|
Di solito l'Applied fornisce un programma tipo "primer express" dove basta inserire le sequenze dei tuoi primer e ti analizza direttamente se formano dimeri, hairpin o altro che ti può falsificare la lettura con il Sybr green come diceva GFPina.
E' utile perchè almeno eviti di sprecare del materiale nel caso in cui il programma ti dice che si possono formare dimeri ecc..
Per quanto riguarda la concentrazione di utilizzo dei primers la ditta consiglia che sia di 200nM, poi in base ai risultati del primo esperimento che farai ti regoli di conseguenza.
 |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|





