| Autore |
Discussione |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 08 marzo 2010 : 13:46:05 Inserito il - 08 marzo 2010 : 13:46:05




|
ciao a tutti,
sono naimi e studio farmacia ,sto preparando l'esame di biologia animale e volevo chiedere alcuni chiarimenti.
ho capito che i telomeri sono le estremità dei cromosomi eucariotici che non codificano per le proteine ma hanno sequenze brevi e esemplici di dna non codificante ripetute più volte. esempio negli uomini 5 TTAGGG-3 ..
adesso non ho capito perchè i telomeri si accorciano e come?
su wikipedia ho trovato questo
I telomeri si accorciano a causa del meccanismo di replicazione del filamento lagging del DNA. Dal momento che la replicazione del DNA non ha inizio dalle estremità ma da varie regioni più centrali di ogni cromosoma e che tutte le DNA polimerasi polimerizzano in direzione 5'->3' (spostandosi in direzione 3'->5'), infatti, il DNA in replicazione presenta un filamento leading ed uno lagging.
Sul filamento leading, la DNA polimerasi può produrre un filamento complementare senza ostacoli, perché procede da 3' a 5'. Al contrario, c'è un problema riguardo al senso 5'->3' che l'enzima dovrebbe prendere sul lagging. Per risolvere questo problema, piccole sequenze di RNA (RNA primer) legano tale filamento e agiscono come innesco, favorendo l'attacco della DNA polimerasi e l'avvio della polimerizzazione. Questo processo genera la formazione dei frammenti di Okazaki.
I frammenti di Okazaki sono processati infine dalla DNA polimerasi, che sostituisce l'RNA dei primers con DNA, e dalla DNA ligasi, che forma il legame fosfodiesterico tra frammenti consecutivi. Questo accade presso tutti i siti del filamento lagging, ma non dove si appaia l'ultimo primer di RNA. [u]In questa regione, infatti, l'RNA viene distrutto da RNAsi, ma non c'è alcuna sostituzione con DNA. Questo genera un continuo processo di accorciamento di queste regioni, che si trovano appunto presso i telomeri.
quello che non capisco è che il filamento guida viene tutto replicato e quello ritardato no? e si perde questo pezzo finale?
|
|
|
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 08 marzo 2010 : 14:40:37 Inserito il - 08 marzo 2010 : 14:40:37




|
| grazie caffey adesso do un'occhiata |
 |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 08 marzo 2010 : 16:59:56 Inserito il - 08 marzo 2010 : 16:59:56




|
caffey ho dato un'occhiata a tutti i messaggi...
però sono confusa ugualmente 
come libro ho il solomon..che è veramente brutto....per cui non sto capendo molto.
Praticamente partendo dalla replicazione, noi abbiamo due filamenti antiparallelo..
quando sintetizziamo il filamento guida, mettiano un innesco in posizione 5' e poi la polimerasi catalizza la sintesi in direzione 5' -> 3'
per sintetizzare il filamento in ritardo : abbiamo bisogno di più inneschi di rna,mettiamo il primo innesco..dopo la dna polimerasi 3 estende il frammento di dna di okazaki da 5 a 3
adesso dobbiamo rimuovere gli inneschi.
la polimerasi 1 comincia a eliminare gli inneschi , il primo ad essere eliminato è quello più vicino all'estremità 3' ...quindi vengono rimossi da 3' a 5 ' .
ma non ho capito questo pezzo che ho trovato in una discussione :
" La DNAP ha bisogno di legarsi a monte del primer da sostituire! Ma come ben sai, a monte del Primer del Lagging strand non c'è niente! Finisce il cromosoma, quindi non può essere sostituito."
prendiamo per esempio questo :
facciamo finta che il primer che sta a 5' sia l'ultimo...
allora la polimerasi rimuove il primer che sta dal lato 3'......per rimuovere questo primer che significa che si lega a monte? che si lega al pezzo blu dal lato 3' ( e quindi va da 3' a 5' ) o che si lega al pezzo blu dal lato rivolto verso 5' ( e va da 5' a 3' ) ?
caffey prima voglio capire questo poi vado avanti , se tu sei disponibile
  |
 |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 08 marzo 2010 : 17:31:06 Inserito il - 08 marzo 2010 : 17:31:06




|
caffey.....
in questo post
Citazione:
Per favore, evita di postare la stessa domanda più volte... Non c'è bisogno!
I Primer vengono rimossi dalla DNAP delta in direzione 3'->5', quindi il primer del leading strand viene normalmente sostituito con DNA, mentre l'ultimo primer del lagging strand non può essere rimosso in quanto a valle non ha DNA a cui si può legare la Pol-delta.
vuoi dire che vengono rimossi i primer che si incontrano a partire da 3' a 5' ?
ma una volta attacato il primer, la dna pol 1 lo rimuove percorrendolo il direzione 5' e 3' ?
e quindi quando dici:
Citazione:
La DNAP ha bisogno di legarsi a monte del primer da sostituire! Ma come ben sai, a monte del Primer del Lagging strand non c'è niente!
vuol dire che l'ultimo primer siccome non presenta verso l'estremità 5' alcun dna...non può subire l'attacco della dna polimerasi ?
|
 |
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
 Inserito il - 08 marzo 2010 : 17:37:25 Inserito il - 08 marzo 2010 : 17:37:25




|
Certo che lo sono. E se non rispondo io risponderà qualcun altro sicuramente, non preoccuparti.
Il primer viene rimosso ma non può essere sostituito come accade per tutti gli altri. La sostituzione del primer avviene come una normale polimerizzazione, ovvero in direzione 5'->3'. Ma come fa la Pol a polimerizzare il tratto che era occupato dal primer se a monte, ovvero in 5' finisce il cromosoma?
La Pol non ha niente a cui legarsi e quindi quel primer viene rimosso e basta, lasciando l'estremità 3' più lunga.
E' più chiaro ora? Se non lo è fammelo presente che insisto!  |
 |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 08 marzo 2010 : 18:02:37 Inserito il - 08 marzo 2010 : 18:02:37




|
grazie caffey mi è chiaro!
grazie davvero !
però adesso mi è venuto un altro dubbio...come sostituire l'innesco di rna al filamento guida.
anche qui verso 5' finisce il cromosoma...
grazie ancora caffey,sei gentilissimo |
 |
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
 Inserito il - 08 marzo 2010 : 18:31:36 Inserito il - 08 marzo 2010 : 18:31:36




|
In entrambe i filamente succede la stessa cosa! Il 5' è più corto del 3'.
Si parla di leading strand e lagging strand quando si prende in considerazione 1 forcella di replicazione. In realtà ambedue i filamenti sono lagging e leading: dipende da come li osservi.
Guarda questa immagine:
Immagine:
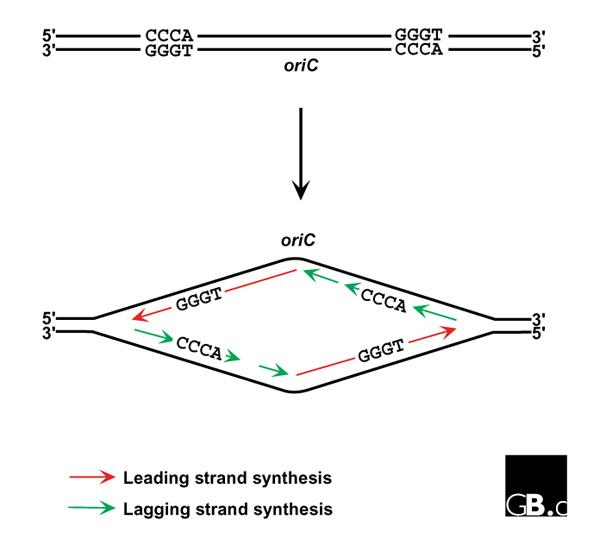
45,34 KB |
 |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 08 marzo 2010 : 19:29:28 Inserito il - 08 marzo 2010 : 19:29:28




|
caffey grazie per l'immagine! :D
cmq l'attività esonucleasica mi confermi che avviene in 3' - 5 ' ? perchè sto cercando da oggi pomeriggio e trovo che avviene in 5'-3 .. |
 |
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
 Inserito il - 08 marzo 2010 : 21:22:54 Inserito il - 08 marzo 2010 : 21:22:54




|
La cosa è molto complessa e differente tra procarioti ed eucarioti. Sono coinvolte endonucleasi (FEN1) e non si sa bene ancora quali polimerasi agiscano in quali processi e come sia la loro attività esonucleasica. A mio parere, l'importante è capire il concetto!
Leggi qui se vuoi chiarirti un po' le idee, comunque: http://en.wikipedia.org/wiki/DNA_polymerase
|
 |
|
|
Naimi
Utente Junior


181 Messaggi |
 Inserito il - 13 marzo 2010 : 19:44:44 Inserito il - 13 marzo 2010 : 19:44:44




|
ti ringrazio di tutto caffey, scusa il ritardo della risposta ma il mio padrone di casa non ha ancora allacciato la connessione 
cmq sei stato gentilissimo |
 |
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
 Inserito il - 13 marzo 2010 : 21:22:50 Inserito il - 13 marzo 2010 : 21:22:50




|
Ma prego! Anche tu sei stata/o gentilissima/o a ringraziare. Ultimamente (non nel forum, per fortuna!) è caduto un po' in disuso, putroppo!
Siamo qui per aiutarci a vicenda!
Buona fortuna per tutto e spero che continuerai a frequentare il forum!
 |
 |
|
| |
Discussione |
|







