| Autore |
Discussione |
|
|
GFAP
Nuovo Arrivato

18 Messaggi |
 Inserito il - 15 luglio 2010 : 21:17:39 Inserito il - 15 luglio 2010 : 21:17:39




|
Salve! Una domanda anzi due:
1) ho fatto il western per il GFAP e per vedere se ho caricato bene ho fatto la beta-actina e è venuta.
Adesso nell'analisi dei dati devo sottrarre i valori della beta-actina da GFAP?
2) Se non sbaglio la Beta actina per esempio per 4 campioni è un loading control quindi teoricamente devono venire tutte bande uguali altrimenti si è sbagliato a caricare vero? O ci sono altre possibilità?
grazie
|
|
|
|
|
cprp77
Nuovo Arrivato

3 Messaggi |
 Inserito il - 22 luglio 2010 : 23:09:38 Inserito il - 22 luglio 2010 : 23:09:38




|
| IO PER NORMALIZZARE GENERALMENTE DIVIDO (non sottraggo!)IL SEGNALE IMMUNOREATTIVO DELLA BANDA DI INTERESSE (NEL TUO CASO GFAP) PER IL SEGNALE DELLA PROTEINA CHE FA DA NORMALIZZANTE (ACTINA)(dello stesso campione). in tal modo puoi comparare l'espressione della GFAP tra i vari campioni che hai caricato nel gel. Ricordati pero' che tale normalizzazione vale solo all'interno del gel che stai analizzando perchè se vuoi comparare gel diversi devi caricare in ciascuno di esso uno standard (es. uno stesso lisato in tutti i gel) e a quel punto dividere il segnale normalizzato dei tuoi campioni per quello dello standard interno. |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 23 luglio 2010 : 08:32:19 Inserito il - 23 luglio 2010 : 08:32:19




|
Concordo, devi dividere non sottrarre.
Citazione:
Se non sbaglio la Beta actina per esempio per 4 campioni è un loading control quindi teoricamente devono venire tutte bande uguali altrimenti si è sbagliato a caricare vero? O ci sono altre possibilità?
Beh, l'idea della normalizzazione è proprio quella di normalizzare le differenze di loading... in teoria vuoi avere tutte e 4 le bande uguali, in pratica è impossibile, quindi normalizzi. Certo è meglio evitare di avere 4 bande completamente differenti l'una dall'altra, ma se un campione è un po' più forte o un po' più debole degli altri non c'è problema. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 23 luglio 2010 : 13:52:30 Inserito il - 23 luglio 2010 : 13:52:30





|
Citazione:
Concordo, devi dividere non sottrarre.
Ne approfitto per cercare di riempire una mia lacuna:
sapete come mai con le bande di WB tutti quanti (me compreso)
dividono per il valore del normalizzatore, mentre nella
real-time PCR tutti quanti sottraggono i Ct del calibratore ?
(per avere il dCt)
|
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 23 luglio 2010 : 14:28:57 Inserito il - 23 luglio 2010 : 14:28:57




|
Il rapporto fra quantità di proteina ed intensità della banda in WB è lineare, quindi quando fai una divisione stai dicendo: il rapporto fra la quantità della mia proteina e dell'actina è x.
Quando guardi il Ct in realtime stai guardando il punto in cui cominci ad avere una crescita esponenziale. Il rapporto fra quantità di DNA e Ct non è lineare ma esponenziale.
Quando dividi due potenze con la stessa base fai effettivamene la differenza fra gli esponenti
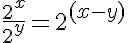 |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
chim2
Utente Attivo
  


2110 Messaggi |
 Inserito il - 23 luglio 2010 : 15:08:12 Inserito il - 23 luglio 2010 : 15:08:12




|
| ma chick nel 1 caso si utilizza uno standard interno no?allora si il rapporto rimane costante sia per quanto riguarda le aree e le concentrazioni |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 23 luglio 2010 : 16:02:15 Inserito il - 23 luglio 2010 : 16:02:15




|
Sì, si usa uno standard interno che si presuppone costante (anche se l'actina non è sempre la scelta migliore...). E proprio perchè rimane costante puoi usare il WB per vedere delle variazioni nell'espressione di un'altra proteina in rapporto allo standard.
PS: qualcuno ha mai testato dei campioni standard a concentrazione crescente in WB? fino a che punto vale la linearità concentrazione/intensità della banda? |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
chim2
Utente Attivo
  


2110 Messaggi |
 Inserito il - 23 luglio 2010 : 16:17:01 Inserito il - 23 luglio 2010 : 16:17:01




|
Citazione:
Messaggio inserito da chick80
Sì, si usa uno standard interno che si presuppone costante (anche se l'actina non è sempre la scelta migliore...). E proprio perchè rimane costante puoi usare il WB per vedere delle variazioni nell'espressione di un'altra proteina in rapporto allo standard.
PS: qualcuno ha mai testato dei campioni standard a concentrazione crescente in WB? fino a che punto vale la linearità concentrazione/intensità della banda?
si può guardare il LOQ del metodo analitico |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 23 luglio 2010 : 16:27:23 Inserito il - 23 luglio 2010 : 16:27:23




|
| Dubito che esista una risposta univoca. Si usano 2 (o più) diversi anticorpi e ci sono diversi metodi di detezione e il campione può contenere milioni di diverse sostanze interferenti... la mia era più che altro una curiosità, ma se qualcuno trova una referenza ben venga! |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFAP
Nuovo Arrivato

18 Messaggi |
 Inserito il - 23 luglio 2010 : 17:39:01 Inserito il - 23 luglio 2010 : 17:39:01




|
| Ho capito grazie! Quindi dividendo. Devo però fare ogni volta la beta-actina per ogni gel dopo il mio anticorpo oppure c'e un modo per paragonare gels diversi dello stesso esperimento? |
 |
|
|
chim2
Utente Attivo
  


2110 Messaggi |
 Inserito il - 23 luglio 2010 : 17:45:35 Inserito il - 23 luglio 2010 : 17:45:35




|
ovviamente sto ragionando in maniera classica,ma sugli interferenti non è cosi' la convalida di un metodo analitico già prende in considerazione tutti gli interferenti di qualsiasi matrice in questo esempio biologica es:
SOP e ICH prevedono varie parametri tra cui il LOD e il LOQ,la Ruggedness e la robustezza,in particolare mi sembra l'ICH prevede la System Suitability ma queste sono fasi finali quindi devi già aver superato la fase di pre convalida passando per i campioni di controllo qualità e tutti i relativi problemi di "interferenti" esempio la concentrazioni dei QC viene fatta a media,bassa e alta concentrazione e la più bassa deve essere minore o uguale 3LOQ per valori medi,mentre per i campioni alti i valori devono essere compresi tra il 75 e il 90% rispetto agli standard di riferimento e nessuna delle concentrazioni scelte deve coincidere con alcun punto della retta di taratura questo è rivolto alla convalida di un metodo specifico che si sta sviluppando.
Per il LOQ il picco dell'analita sia identificabile,riproducibile con una precisione del 20% e un'accuratezza dall'80 al 120%.
La linearità del metodo viene determinata analizzando una serie di campioni tipo a 7 concentrazioni diverse,si raccomanda sempre,almeno le agenzie internazionali che se ne occupano consigliano sempre di utilizzare la regressione più semplice o almeno senza coefficienti pesati,in alternativa se deve giustificare se si usano altri tipi di regressione..
cmq secondo queste agenzie
-la deviazione del limite di quantificazione deve essere minore o uguale il 20% rispetto alla concentrazione nominale
-che la deviazione degli standard sia minore o uguale il 15 %
-che almeno 4 fra 6 standard differenti devono rispettare questi criteri
-ovviamente che il coefficiente di correlazione sia maggiore o uguale al 95%
poi si passa a determinare il range.
Ritornando al LdR è fissato a una distanza di 3 "sigmab"
la regione che va dal 3 sigmab al 10 sigmab rappresenta la rivelabilità del tuo analita
al 10 sigmab staimo sul LOQ
alla fine il limite di linearità corrisponde a quella concentrazione che corrisponde a un segnale ottenuto inferiore al 5%,puoi trovare autori che ti dicano anche il 3% ovviamnete cosi' non tieni conto della variabilità delle misure ma per convalidare alla fine diverse agenzie tengono conto di parametri finali come la system suitability che quindi poi va a considerare anche tutto l'apparato strumentale nel suo complesso,perchè oltre agli interferenti va considerato anche l'apparato strumentale nel suo complesso cioè viene considerato come un unico organico..quindi per gli strumenti ti da delle indicazioni sui segnali se questi non vanno bene questa agenzia ti scarta il metodo analitico e te lo rimanda a casa..
questo si fa in maniera classica in campo analitico e le agenzie di solito danno questi parametri,in ambito accademico sicuramente ce ne sono altri che si stanno studiando,ma in termini di lavoro so quelli 
P.S.normalizzare un'area o un'altezza non è la stessa cosa di usare il metodo dello standard interno |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 23 luglio 2010 : 18:41:27 Inserito il - 23 luglio 2010 : 18:41:27




|
Citazione:
Ho capito grazie! Quindi dividendo. Devo però fare ogni volta la beta-actina per ogni gel dopo il mio anticorpo oppure c'e un modo per paragonare gels diversi dello stesso esperimento?
No, devi rifare l'actina ogni volta.
@chim2, quello che dici è giustissimo, ma non è applicabile nel caso di un Western blot, che è un metodo semi-quantitativo. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
chim2
Utente Attivo
  


2110 Messaggi |
 Inserito il - 23 luglio 2010 : 18:53:27 Inserito il - 23 luglio 2010 : 18:53:27




|
e allora non so la risposta  |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 23 luglio 2010 : 21:04:24 Inserito il - 23 luglio 2010 : 21:04:24





|
Tornando alla tua domanda chick:
Citazione:
Messaggio inserito da chick80
PS: qualcuno ha mai testato dei campioni standard a concentrazione crescente in WB? fino a che punto vale la linearità concentrazione/intensità della banda?
Dipende dal tipo di anticorpo, come si lega alla proteina, se è marcato direttamente o se utilizzi un secondario, il sistema di rivelazione che utilizzi... le variabili sono tante. Puoi trovare una zona dove hai linearità per quella determinata proteina e quel determinato anticorpo/ metodo di rilevazione, ma non sarà lo stesso per tutte le proteine presenti nel tuo campione.
Comunque è stato fatto, ad es. vedi da questo articolo:
http://www.nature.com/npp/journal/v32/n4/full/1301178a.html
la figura supplementare 1:
http://www.nature.com/npp/journal/v32/n4/extref/1301178x2.pdf (immagine)
http://www.nature.com/npp/journal/v32/n4/extref/1301178x1.doc (didascalia)
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 24 luglio 2010 : 09:05:42 Inserito il - 24 luglio 2010 : 09:05:42




|
| Era proprio quello che cercavo! :) (non ho fatto un solo WB negli ultimi 5 anni e non è nei miei piani di farne prossimamente... ma è sempre buono a sapersi) |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
| |
Discussione |
|