| Autore |
Discussione |
|
|
cry87
Utente Junior


Città: milano

127 Messaggi |
 Inserito il - 25 novembre 2010 : 16:45:35 Inserito il - 25 novembre 2010 : 16:45:35




|
Ciao,mi aiutate con questo esercizio? Si tratta di trovare una strategia di clonaggio adatta.
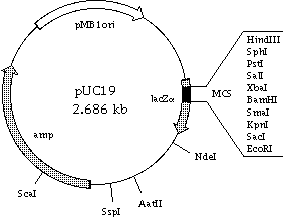
La sequenza sotto riportata appartiene ad un gene del batterio Acinetobacter baumannii che codifica per un recettore del ferro. La proteina è considerata di interesse biotecnologico. Disegnate una semplice strategia di clonaggio per l’espressione del gene in E. coli, utilizzando il plasmide pUC19 rappresentato in figura.
caacaatttcagtcaaagctgataaggaaagcagtttaaaacaagaagtggggcaagcat
caagtgcaactaaaggacttATGCAATTAAAAGATGTCCCTCAAATTGTAAACGTTGTAC
CCAAACAAGTTTTAAGAGAACAGACAGTTACCTCTATGCAAGGTGCCTTGCAAAACGTTG
CAGGTTTAAGCTTTAGTGTAGGAGATGGACAACGTGATCAGGTCATGATTCGTGGTTTCT
CTGCAATCACCGATAACTATGTAGATGGTATTCGCGATGACGCATTGTATTTTCGCGATA
……………………………………………………………………………………………………………………………………………………………
……………………………………………………………………………………………………………………………………………………………
……………………………………………………………………………………………………………………………………………………………
TAGCAGGTGAGCAAAAAACCAAAGGTGTTGAAATTTCATTAGCAGGTCAGTTGACCGATC
AATTATCAGTATTGGCCGGTTATTCGTATATGGATGGTAAGATTGAAAAATCGGCTATTG
GTTTTACAGGCAATCATTCTGCGCTCACGCCAAACAACACTGCTAACTTGTGGTTAAAAT
ATCAAATTAATGATCATTGGTATGCAGCTGTGGGTGGTCGTGGTGAATCATCACGTTTTA
GTGCGCCAGACAACAAAAACGTTTTACCGGGATATGCAGTTGTAAATGCGGCGTTAGGTT
ATCAAAGTGAACGTTATGATGTGAACTTAAACTTAAATAACTTGTTTGATCGCGATTACT
TTGTGTCAGGTCATAGTGGAGCAAATGATTCGAACATGATGGGCGATCCTTTAAATGCTC
AAGTTGCACTTCGCTATCGTTTTTAAttaaaaaaactctaaaaagctcatcatttcgatg
Agcttttttatttgtcgtgtaggatatagatagtattaaaaggttttgttctgataggat
Immagine:
2,71 KB
grazie mille!!
cry
|
|
|
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 26 novembre 2010 : 11:50:58 Inserito il - 26 novembre 2010 : 11:50:58




|
Piu' che altro si tratta di scegliere i siti di restrizione piu' utili, in modo da assicurarti che la direzione in cui la tua proteina X viene clonata sia la stessa di quella di LacZ. Immagino infatti che per l'espressione in E. coli utilizzerai il promotore che dirige l'espressione dello stesso LacZ (IPTG-inducible). Dal momento che il polylinker è all'interno di LacZ (o meglio della parte che codifica i primi amminoacidi N-terminali di LacZ) dovrai fare attenzione che il tuo gene sia in frame con questi primi codoni(il risultato sarà quello di avere una proteina chimerica con qualche aa N-terminale derivante da LacZ e poi la tua proteina stessa).
Il tutto si presta al saggio blue/white con l'X-gal quindi saprai quali colonie hanno l'inserto e quali no. |
 |
 |
|
|
cry87
Utente Junior


Città: milano

127 Messaggi |
 Inserito il - 26 novembre 2010 : 12:52:56 Inserito il - 26 novembre 2010 : 12:52:56




|
ciao,grazie mille x la risposta ma ho un paio di dubbi! sicuramente banali!
-come scelgo i siti di restrizione?
-devo disegnare i primers?
|
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 26 novembre 2010 : 15:02:34 Inserito il - 26 novembre 2010 : 15:02:34




|
1)I siti di restrizione li devi scegliere in base a frame e direzionalità (ne scegli per esempio due diversi, in modo che la orf del gene X verrà clonata nella direzione giusta, e tali che il frame sia mantenuto).
2)e' possibile fare un passaggio in cui amplifichi il gene X tramite pcr. in tal caso i primers dovranno contenere i siti di restrizione prescelti.
Es:
hind III LacZ----->EcoRI tagli con HindIII ed EcoRI
HindIII geneX ---->ecoRI anche qui tagli con Hind ed Eco
ligation etc etc |
 |
 |
|
|
RM
Nuovo Arrivato

115 Messaggi |
 Inserito il - 26 novembre 2010 : 15:06:25 Inserito il - 26 novembre 2010 : 15:06:25




|
Ciao
Per i siti di restrizione usi quelli del MCS, che ovviamente non tagliano anche la tua sequenza e che saranno contenuti anche nei tuoi primers (da disegnare).
Volevo anche aggiungere che il puc19 è un vettore di clonaggio, non di espressione. Il saggio blue/white si basa sul fatto che, se viene inserito un inserto, il gene per lacz viene inattivato e non idrolizza l' x-gal. Quindi se le colonie sono positive saranno bianche, viceversa blu. Non è quindi una questione di espressione chimerica o di fusione con lacz.
Ciao |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 26 novembre 2010 : 15:30:43 Inserito il - 26 novembre 2010 : 15:30:43




|
si' ma il gene lacZ lo devi indurre, tramite l'aggiunta di IPTG. tant'è che se aggiungi X-gal a piastre senza iptg, le colonie saranno tutte bianche poichè lacZ nn è espresso. Quindi immagino che questo lacZ (che alfa complementa quello difettoso del ceppo di E. coli che si usa in tal caso) sia regolato da un promotore iptg inducibile. Quindi se tu inserisci il tuo gene X nel multicloning site potresti indurre poi l'espressione di questo aggiungendo IPTG al mezzo di coltura. O sbaglio? Cosa che tra l'altro sarebbe ottimale nel caso in cui la proteina si riveli tossica per i batteri (ne induci l'espressione solo quando stai al massimo della curva di crescita...).
Riguardo al frame intendevo dire che siccome il mcs è all'interno di lacZ se vuoi esprimere la tua proteina essa dovrà essere in frame con l'inizio di di lacZ.
|
 |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
|
|
RM
Nuovo Arrivato

115 Messaggi |
 Inserito il - 26 novembre 2010 : 15:58:39 Inserito il - 26 novembre 2010 : 15:58:39




|
Quello che dici è corretto.
Volevo solo sottolineare come però lo screening blue/white non si basa necessariamente sul clonaggio in frame con lacz (cioè si puo fare anche se l'inserto da clonare non è in frame) |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 26 novembre 2010 : 16:06:04 Inserito il - 26 novembre 2010 : 16:06:04




|
| ah beh certo lo screening blu/white dipende solo dal fatto che "distruggi" la lacZ. |
 |
 |
|
|
cry87
Utente Junior


Città: milano

127 Messaggi |
 Inserito il - 26 novembre 2010 : 16:07:57 Inserito il - 26 novembre 2010 : 16:07:57




|
| grazie mille a tutti e 2!!!!!! |
 |
|
|
cry87
Utente Junior


Città: milano

127 Messaggi |
 Inserito il - 28 novembre 2010 : 14:31:36 Inserito il - 28 novembre 2010 : 14:31:36




|
Ho riletto tutte le vostre risposte e se prima avevo capito ora sono in panico perchè non ho più pallida idea di come disegnare i primers!! Devo disegnare sia il reverse che il forward? oddio |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 28 novembre 2010 : 16:38:01 Inserito il - 28 novembre 2010 : 16:38:01




|
non capisco dove sia la tua difficoltà. Ti disegni due primer, il forward contenente un sito di restrizione, il reverse con un sito di restrizione diverso. Per esempio potresti usare il forward con HindIII e il reverse con BamHI. Questo ti assicura che la ORF del tuo gene sarà nell'orientamento giusto rispetto al promotore lacZ (l'orientamento è indicato dalla freccia di lacZ nella mappa).Riguardo al frame mi riferivo al fatto che se tu devi esprimere questa proteina in E. coli proprio con il pUc19, allora è necessario che il tuo prodotto sia in frame con LacZ, visto che il multi cloning site (MCS) è interno a lacZ (altrimenti se non è in frame potresti inserire codoni di stop prematuri, o cambiare la sequenza ecc...ti viene fuori un casino, no?). Certo è difficile progettare un primer che sia in frame con lacZ se non conosci la sequenza del MCS, quindi forse è un particolare che nn ti viene richiesto dall'esercizio.
http://www.mobitec.com/int/products/bio/04_vector_sys/stcl_v.html
Spero di nn averti confuso le idee...
|
 |
 |
|
|
cry87
Utente Junior


Città: milano

127 Messaggi |
 Inserito il - 07 dicembre 2010 : 18:10:52 Inserito il - 07 dicembre 2010 : 18:10:52




|
grazie mille! ora ho filalmente capito!!! |
 |
|
|
cry87
Utente Junior


Città: milano

127 Messaggi |
 Inserito il - 16 dicembre 2010 : 16:54:33 Inserito il - 16 dicembre 2010 : 16:54:33




|
SpemannOrganizer in questo esercizio non devo creare una proteina di fusione quindi il frame NON deve essere mantenuto |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 16 dicembre 2010 : 17:54:14 Inserito il - 16 dicembre 2010 : 17:54:14




|
| perfetto. Quindi il compito è + semplice :) |
 |
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 24 dicembre 2010 : 00:31:46 Inserito il - 24 dicembre 2010 : 00:31:46





|
Secondo me il frame deve essere proprio evitato, sopratutto per inserti di piccole dimensioni, si corre il rischio di ottenere dei falsi negativi.
Se il frammento è piccolo (fino a 400-500 pb) e si inserisce in frame, è possibile che non dia noia all' espressione di LacZ e si ottengano colonie blu.
Col p-Gem T easy ad esempio, non si può controllare come si inserisce l'inserto, e se il frammento è picciono può capitare quello che ho detto sopra. ( a me è successo più volte di quante me ne potevo aspettare 0_0)
|
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 24 dicembre 2010 : 08:34:55 Inserito il - 24 dicembre 2010 : 08:34:55




|
| ma infatti il frame nn importa se deve essere clonato semplicemente all'interno del vettore. Il problema all'inizio della questione era "Disegnate una semplice strategia di clonaggio per l’espressione del gene in E. coli" cioè io personalmente avevo inteso che questo gene doveva essere espresso in E. coli. In tal caso invece bisognerebbe rispettare il framevisto che il promotore è lo stesso che regola LacZ e visto che il polylinker è all'interno di lacZ. Cry87 ha pero' successivamente detto che questo particolare nn era richiesto dall'esercizio. |
 |
 |
|
|
5ringsman
Nuovo Arrivato

11 Messaggi |
 Inserito il - 04 novembre 2011 : 12:35:45 Inserito il - 04 novembre 2011 : 12:35:45




|
| Ciao a tutti... Ho trovato interessante la discussione .... Io ho lo stesso problema con l'aggiunta che devo esprimere la proteina e quindi ho bisogno che sia in frame col promotore. Il mio vettore è il pET28... Esite un qualche tool bioinformatico che mi dice "se uso questo o quel sito di restrizione per ligare l'inserto, la proteina codificata all'interno è in frame?" grazie mille |
 |
|
| |
Discussione |
|







