| Autore |
Discussione |
|
|
Vars
Nuovo Arrivato

44 Messaggi |
 Inserito il - 22 febbraio 2011 : 09:15:40 Inserito il - 22 febbraio 2011 : 09:15:40




|
|
Ciao, volevo chiedere una cosa e nn so se sia una domanda stupida o meno, cmq ho un dubbio...se digerisco un plasmide con due diversi enzimi di restrizione, ho minor rischio che si creino molecole aberranti durante il mio esperimento di clonaggio giusto? la mia domanda è : l'inserto devo digerirlo con gli stessi due enzimi di restrizione che ho usato per il vettore? e per quanto riguarda il mio vettore, che tipo di molecola otterrò? cioè ottengo una molecola lineare? e quali saranno le estremità di questa molecola? potreste spiegarmelo graficamente? ve ne sarei grata!
|
|
|
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 22 febbraio 2011 : 12:20:13 Inserito il - 22 febbraio 2011 : 12:20:13




|
Si, sempre che le estremità non siano compatibili, anche solo parzialmente. Per questo di norma si defosfata il vettore. In assenza di 5'P infatti la ligasi non puoi catalizzare la richiusura del plasmide, detta anche ricircolarizzazione. Si usa tale termine perché a seguito del taglio, con uno o nel caso da te citato con due enzimi di restrizione, si crea un vettore lineare. Si suol dire perciò che il vettore è stato linearizzato, le cui estremità saranno ovviamente:
- da un lato l'estremità data da un enzima di restrizione;
- dall'altro lato avrai un'estremità frutto del taglio con il secondo sito di restrizione.
Affinché il frammento si leghi al vettore dovrà perciò avere estremità complementari alle estremità del vettore lineare.
Come immagine non è il massimo, ma fai riferimento a questa:
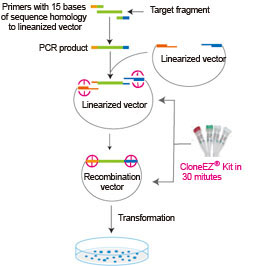
Non badare al primo passaggio in PCR: questo viene performato per aumentare l'efficienza di clonaggio e soprattutto per introdurre nel frammento gli appropriati siti di restrizione alle estremità, grazie all'uso di primer specificamente disegnati per tale scopo. In tale modo si evita il clivaggio all'interno del frammento, che viene scongiurato tramite metilazione dello stesso.
|
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 22 febbraio 2011 : 12:37:14 Inserito il - 22 febbraio 2011 : 12:37:14




|
Citazione:
Messaggio inserito da 0barra1
...In tale modo si evita il clivaggio all'interno del frammento, che viene scongiurato tramite metilazione dello stesso.
Cosa intendi dire? |
 |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 22 febbraio 2011 : 14:38:54 Inserito il - 22 febbraio 2011 : 14:38:54




|
| Che onde evitare tagli all'interno del DNA da clonare, si può metìlarlo e quindi aggiungere alle estremità del frammento protetto dal taglio il sito di restrizione desiderato, sicuri così ch la endonucleasi agirà solo lì. |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 22 febbraio 2011 : 14:53:33 Inserito il - 22 febbraio 2011 : 14:53:33




|
ma in pratica, come fai a metilarlo?nella pcr nn ci sono enzimi che metilano, nella pratica non ho mai sentito della possibilità di metilare il proprio DNA/costrutto APPOSITAMENTE per evitare la digestione con i propri enzimi. Poi per carità forse è possibile metter su una reazione apposita...
Nella mia realtà di laboratorio le uniche fonti di metilazione del DNA sono i batteri. Pertanto, onde evitare di digerire accidentalmente il proprio costrutto, si scelgono semplicemente siti di restrizione (e quindi enzimi) che nn stanno all'interno del nostro costrutto. |
 |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 23 febbraio 2011 : 03:37:20 Inserito il - 23 febbraio 2011 : 03:37:20




|
Partiamo dalle cose di cui sono più sicuro: ovviamente non esiste una metilasi da PCR, come noti tu. Tuttavia questo ostacolo è superato dall'impiego nella miscela di reazione di metilcitosine e metiladenine. Il DNA amplificato si trova quindi metilato, posso quindi applicare per esempio dei linker con il sito di restrizione e poi tagliare.
Per quanto riguarda la metilazione diretta, le tue perplessità mi lasciano un po' spiazzato. Nel senso che mi son trovato da poco a dare un esame inerente a queste tematiche e dal corso mi è sempre stato proposto con grande naturalezza la possibilità, per es., di metilare i frammenti prima di legarvi un linker e quindi clonare. Da ciò avevo supposto che si metilasse in vitro, prima di ligare. Dovrei ricontrollare su appunti e testi, attendo chiarimenti. Purtroppo con l'ultimo pesantissimo esame alle porte le mie risposte arriveranno dilazionate nel tempo.
 |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 23 febbraio 2011 : 04:40:38 Inserito il - 23 febbraio 2011 : 04:40:38




|
| ma no sai,può essere che sia una metodica recente di cui sono 'unaware',all'oscuro.posso solo dirti che di clonaggi ne ho fatti davvero tanti,anche nn tradizionali,vedi gateway.ma niente metilcitosine nella pcr o metilazione diretta. solo un'attenta pianificazione prima di disegnare i primer.tra l altro diversi enzimi sn insensibili alla metilazione quindi sarebbe una strategia circoscritta per sua stessa natura. cmq mi informerò xche la cosa mi interessa. Studi di notte o,cm me,hai problemi a dormire?:) |
 |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 23 febbraio 2011 : 12:07:58 Inserito il - 23 febbraio 2011 : 12:07:58




|
Studio di notte e ho problemi a dormire  . Cavolo se però hai postato tardi! O presto... . Cavolo se però hai postato tardi! O presto...
Comunque non sono ancora riuscito a controllare appunti vai, tuttavia appena possibile ti aggiornerò.
 |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 23 febbraio 2011 : 12:16:19 Inserito il - 23 febbraio 2011 : 12:16:19




|
good, la cosa è interessante. la cosa che mi interessa sapere è se è una pratica usata per evitare digestioni interne al proprio inserto (di cui magari nn si conosce la sequenza - io in tal caso farei un ta cloning o topo o gateway - ). Poi so che ci sono metodiche per metilare sequenze di dna, per esempio per studiare methyl binding protein o metilazione e attività del promotore ecc.Nella mutagenesi sito diretta invece si sfrutta il contrario: si metila il vettore parentale (tramite batteri o in vitro) e si digerisce con un enzima di restrizione specifico per dna metilato, lasciando il mutato che invece nn è metilato...
Ciao e un grazie in anticipo |
 |
 |
|
| |
Discussione |
|







