Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 18 maggio 2011 : 13:51:05 Inserito il - 18 maggio 2011 : 13:51:05




|
Ciao ragazzi, sul mio libro di biologia molecolare in un paragrafo intitolato "RFLPs and SNPs can be used for genetic mapping" compare una figura che non sono riuscito a 'decifrare'  anche perchè nel testo praticamente nemmeno la menzionano, e la sua didascalia è abbastanza povera. La figura originale non l'ho trovata, ma ve la propongo per come sono riuscito a copiarla (è un po' brutta, ma le informazioni e lo schema sono proprio gli stessi). anche perchè nel testo praticamente nemmeno la menzionano, e la sua didascalia è abbastanza povera. La figura originale non l'ho trovata, ma ve la propongo per come sono riuscito a copiarla (è un po' brutta, ma le informazioni e lo schema sono proprio gli stessi).

Restriction marker is 30 map units from eye color marker
[questa è la didascalia]: A restriction polymorphism can be used as a genetic marker to measure recombination distance from a phenotypic marker (such as eye color). The figure simplifies the situation by showing only the DNA bands corresponding to the allele of one genome in a diploid. (le strisce in violetto sono corse su gel, ibridizzate con una sonda dopo un southern immagino - i cerchi indicano il colore degli occhi di ogni individuo, le prime due sono i genitori e le quattro sotto sono la progenie)
Ora, capisco che si sta tentando di capire la distanza di mappa tra i due marker, uno fenotipico (colore occhi) e l'altro basato su di un polimorfismo di restrizione, in base alle frequenze di ricombinazione.
Ma la figura non sono riuscito ad interpretarla. Innanzitutto il carattere colore occhi in Drosophila è legato al sesso, questo va tenuto in considerazione? E i genotipi parentali quali sono? Non viene specificato il sesso di ogni genitore...immagino non serva. 
Sapreste darmi una mano a capire la figura e i dati allegati per favore?

|
|
|
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 18 maggio 2011 : 20:40:53 Inserito il - 18 maggio 2011 : 20:40:53




|
Avete desistito tutti? 
Vi prego, un aiutino! Se vi serve qualche info in più cercherò d'essere più chiaro. |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 18 maggio 2011 : 21:48:27 Inserito il - 18 maggio 2011 : 21:48:27





|
E' una figura "semplificata" che secondo me alla fine complica le cose in effetti!
Considerando solo il polimorfismo tu hai 2 alleli:
- allele 1: una sola banda (perché l'enzima non taglia)
- allele 2: 2 bande (perché l'enzima taglia)
Avendo un genitore 2,2 e l'altro 1,1 tu ti aspetteresti ovviamente dei figli 1,2 (quindi con 3 bande), che qua non vedi!
Quelli non sono i figli!
Penso che quello che hai scritto tra parentesi, sia una deduzione tua:
Citazione:
...le prime due sono i genitori e le quattro sotto sono la progenie)
La figura ti dice semplicemente:
1) nei genitori ho che:
- il carattere "colore rosso" è associato con l'allele 2
- il carattere "colore bianco" è associato con l'allele 1
2) nei "figli" osservo che:
- nel 35% dei casi il carattere "colore rosso" è ancora associato con l'allele 2
- nel 35% dei casi il carattere "colore bianco" è ancora associato con l'allele 1
MA
- nel 15% dei casi il carattere "colore rosso" è INVECE associato con l'allele 1
- nel 15% dei casi il carattere "colore bianco" è INVECE associato con l'allele 2
Quindi ho il 30% di ricombinanti tra polimorfismo e "colore occhi" e questo mi dice che i due marker sono a 30u.m.
N.B. ho messo "figli" tra virgolette perché quelli non possono essere i figli di quei genitori come dicevo prima, ovviamente per poter osservare i ricombinanti tu dovresti avere un genitore eterozigote.
Chiamando: A= allele occhi rossi e a = allele occhi bianchi
1 = allele polimorfismo 1 (1 banda) e 2 = polimorfismo 2 (2 bande)
Potresti avere questo individuo etereozigote:
a1/A2
che produce Gameti parentali: a1 e A2
e Gameti ricombinanti: a2 e A1
se hai: 35% a1 35% A2 e 15% a2 e 15% A1
allora i due marker stanno a 30u.m.
magari se la figura l'avessero disegnata così sarebbe stata più chiara! |
 |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 18 maggio 2011 : 22:37:18 Inserito il - 18 maggio 2011 : 22:37:18




|
Grazie mille, chiarissima come sempre. Allora la figura era più semplice di quanto pensassi; mi ha tratto in inganno quella freccia. Quindi la parte inferiore della figura altro non è che la schematizzazione dei gameti prodotti con le loro relative frequenze tenuto conto degli eventi di ricombinazione tra i due loci.
Ora mi rimane un altro dubbio: quando si parla di un polimorfismo di restrizione associato (linked) ad un particolare fenotipo, e in particolare in procedure di diagnosi, ad una malattia genetica, si intende quindi che quel RFLP è così vicino al sito mutato nella malattia praticamente da non poterne essere mai separato da eventi di crossing over, giusto? A quanto ho capito quindi la logica sarebbe quella di confermare, diagnosticare o addirittura predire una malattia genetica se si trova nel DNA dell'individuo il RFLP "linkato" al locus mutato nella malattia stessa (quindi usare il RFLP come marker).
Mi rimane però strano il fatto di associare così strettamente le due cose, cioè in fondo il SNP di una malattia e un RFLP vicino (sempre che non coincidano ovviamente) sono due eventi, si associati tra loro a livello cromosomico, ma indipendenti e non interagiscono per nulla tra loro. Quindi tutto questo ambaradam si basa sulla casistica di popolazione? Cioè che la maggior parte degli individui che hanno uno dei due polimorfismi presenterebbero anche il secondo... se fosse così di certo ci sarebbe ben poco, no?  |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 19 maggio 2011 : 00:49:53 Inserito il - 19 maggio 2011 : 00:49:53





|
Citazione:
Messaggio inserito da Geeko
Quindi la parte inferiore della figura altro non è che la schematizzazione dei gameti prodotti con le loro relative frequenze tenuto conto degli eventi di ricombinazione tra i due loci.
No nella figura non sono indicati i gameti, quella alla fine era solo una mia considerazione, ovvero se avessero messo i figura un individuo eterozigote e i suoi gameti avresti avuto quella stessa situazione e la figura sarebbe risultata chiara. (infatti l'avresti capita)
Cosa sia in realtà rappresentato in figura non si capisce! Hanno cercato di semplificare le cose mettendo in realtà una cosa che non ha molto senso e scrivendo: "The figure simplifies the situation by showing only the DNA bands corresponding to the allele of one genome in a diploid." Senza dare ulteriori spiegazioni.
Comunque sia il senso è quello che ti ho spiegato.
(la figura originale comunque c'è in internet, è qui: http://books.google.it/books?id=kEtMNqEu7MYC&pg=PA76)
Citazione:
Messaggio inserito da Geeko
Ora mi rimane un altro dubbio: quando si parla di un polimorfismo di restrizione associato (linked) ad un particolare fenotipo, e in particolare in procedure di diagnosi, ad una malattia genetica, si intende quindi che quel RFLP è così vicino al sito mutato nella malattia praticamente da non poterne essere mai separato da eventi di crossing over, giusto?
Sì si utilizzano polimorfismi molto vicini al "gene malattia", ovviamente più vicino è meglio è perché ci sarà meno la P di avere ricombinazione.
Citazione:
Messaggio inserito da Geeko
A quanto ho capito quindi la logica sarebbe quella di confermare, diagnosticare o addirittura predire una malattia genetica se si trova nel DNA dell'individuo il RFLP "linkato" al locus mutato nella malattia stessa (quindi usare il RFLP come marker).
Mi rimane però strano il fatto di associare così strettamente le due cose, cioè in fondo il SNP di una malattia e un RFLP vicino (sempre che non coincidano ovviamente) sono due eventi, si associati tra loro a livello cromosomico, ma indipendenti e non interagiscono per nulla tra loro. Quindi tutto questo ambaradam si basa sulla casistica di popolazione? Cioè che la maggior parte degli individui che hanno uno dei due polimorfismi presenterebbero anche il secondo... se fosse così di certo ci sarebbe ben poco, no? 
No su questa parte finale non ci siamo...
tu puoi fare una analisi di questo tipo all'interno di una famiglia, ma non generalizzare su tutta la popolazione. Mi spiego meglio.
L'unica cosa che sai è che il carattere "colore occhio" è in linkage con l'RFLP 1-2, cioè quei due pezzi di DNA sono vicini.
Però non è affatto vero che l'occhio rosso sia SEMPRE associato all'allele 2 e l'occhio bianco all'allele 1. Puoi avere tutte le combinazioni possibili.
Quello che fai però è analizzare una singola famiglia e vai a vedere i componenti di quella famiglia che hanno occhio bianco che RFLP hanno e vedi ad es. che "in quella famiglia" i "malati" (occhio bianco) hanno l'allele 1, quindi ne deduci che se nasce una drosophila con l'allele 1 avrà verosimilmente occhio bianco. Però potresti avere anche un'altra famiglia in cui il bianco è associato al 2, tutti i componenti della famiglia con occhio bianco hanno l'allele 2, quindi se nasce una Drosophila con allele 1 molto probabilmente avrà occhi rossi a differenza di quella della prima famiglia.
Insomma il marcatore ti permette in questo caso di seguire "indirettamente" la trasmissione del carattere "malattia" che è quello che ti realmente ti interessa, ma non è la stessa cosa.
In genere si analizzano gli alberi genealogici di una famiglia e hai ad es. una cosa del genere:
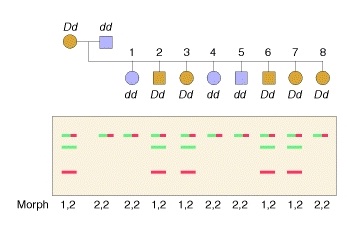
(immagine presa da: http://www.ncbi.nlm.nih.gov/books/NBK21320/
dove trovi anche una spiegazione) |
 |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 19 maggio 2011 : 08:10:24 Inserito il - 19 maggio 2011 : 08:10:24




|
Allora i miei dubbi erano fondati! A quanto letto dal libro però non veniva mai specificato che si trattasse di analisi a raggio così ristretto fatte quindi "ad hoc" per un gruppo di persone imparentate. Ora ha effettivamente più senso 
Appena ho tempo leggo tutta quella pagina dell'NCBI
Grazie! |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-18 MolecularLab.it |
 |
|
|




 anche perchè nel testo praticamente nemmeno la menzionano, e la sua didascalia è abbastanza povera. La figura originale non l'ho trovata, ma ve la propongo per come sono riuscito a copiarla (è un po' brutta, ma le informazioni e lo schema sono proprio gli stessi).
anche perchè nel testo praticamente nemmeno la menzionano, e la sua didascalia è abbastanza povera. La figura originale non l'ho trovata, ma ve la propongo per come sono riuscito a copiarla (è un po' brutta, ma le informazioni e lo schema sono proprio gli stessi).


