| Autore |
Discussione |
|
|
moni89
Nuovo Arrivato


60 Messaggi |
 Inserito il - 27 giugno 2011 : 12:27:00 Inserito il - 27 giugno 2011 : 12:27:00




|
Ho cercato sul forum in altre discussioni e ho trovato che la fosfatasi alcalina serve ad impedire che il vettore si richiuda su se stesso prima di inglobare l'inserto. Sulle dispense del prof però dice che la fosfatasi alcalina serve ad impedire la ligazione di più inserti e va utilizzata sull'inserto, non sul vettore...
In pratica dice che per impedire la ligazione di più inserti si tratta il cosmide (vettore) con due enzimi di restrizione diversi e l'inserto con fosfatasi... questo non mi è chiaro!
|
|
|
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 27 giugno 2011 : 12:44:30 Inserito il - 27 giugno 2011 : 12:44:30




|
| Non sono un esperto in materia, ma ragionandoci semplicemente un po', credo che sia necessario aggiungere la fosfatasi al campione d'inserto per impedire che si formino delle serie di inserti concatenati. Ma in fondo lo stesso problema dovrebbe averlo anche il vettore; anche se tagliato con due enzimi diversi, questo non impedisce che si formino di nuovo delle catene di più vettori (di solito il doppio taglio con due enzimi permette di ottenere un'orientamento fisso dell'inserto). Quindi non vedo perchè non si debba trattare anche il vettore con la fosfatasi. |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 27 giugno 2011 : 13:09:12 Inserito il - 27 giugno 2011 : 13:09:12




|
Solitamente si tratta il vettore con la fosfatasi, di modo tale da ridurre il rischio di richiusura su se stesso del vettore, oltre che la formazione di eventuali, più rari, concatameri di vettore. Al contrario il DNA non è trattato con fosfatasi, altrimenti sarebbe impossibile ligare.
La strategia cambia qualora si intenda realizzare una libreria di cDNA per mappare magari un genoma od una sua regione. Dal momento che si è particolarmente interessati alla sua rappresentatività (la libreria deve essere fedele all'originale), non è tollerabile che frammenti di cDNA diversi siano legati tra loro accidentalmente nella reazione di ligazione. In questo caso, infatti, finiresti per vedere adiacenti sequenza di DNA che in realtà potrebbero essere separate nel genoma. E dunque sono i frammenti da clonare ad esser fosfatati, anziché i vettori. |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
moni89
Nuovo Arrivato


60 Messaggi |
 Inserito il - 27 giugno 2011 : 13:28:08 Inserito il - 27 giugno 2011 : 13:28:08




|
| Quindi in pratica si tratta l'inserto con fosfatasi per impedire che diversi inserti si leghino tra loro? Ho capito bene? |
 |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 27 giugno 2011 : 14:03:38 Inserito il - 27 giugno 2011 : 14:03:38




|
A quanto detto da Obarra1 è proprio il vettore ad essere trattato con fosfatasi, e non l'inserto.
Il concetto è, ditemi se sbaglio, che per far avvenire la ligazione, è necessario un nucleoside trifosfato ad ogni estremità per formare un nuovo legame fosfodiesterico. Quindi, almeno da punto di vista della reazione in sé, è indifferente che questo sia sull'inserto, o sul vettore, giusto?

Ma in ogni caso le emieliche da "saldare" sono sempre due, quindi non sarebbe necessario avere 'fosfati' disponibili su entrambi i frammenti da ligare (vettore e inserto)? |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 27 giugno 2011 : 14:30:12 Inserito il - 27 giugno 2011 : 14:30:12




|
Citazione:
Messaggio inserito da moni89
Quindi in pratica si tratta l'inserto con fosfatasi per impedire che diversi inserti si leghino tra loro? Ho capito bene?
Si, hai capito bene. Si adotta tale approccio per evitare di co-clonare nello stesso vettore due o più inserti, di modo che i coni siano sovrapponibili (contig di cloni) correttamente.
@Geeko:
La situazione è la seguente, in seguito a defosfatazione del vettore:

Il trattamento come vedi permette di saldare solo 2 dei 4 nick (e nello specifico il nick di destranel filamento superiore, il nick di sinistra in quello inferiore), il che riduce l'efficienza di trasformazione. Questo perché i plasmidi hanno una probabilità ben maggiore di trasformare batteri se i plasmidi stessi sono superavvolti. La procedura comunque ha successo e i nick vengono quindi riparati dai sistemi di riparazione del DNA della cellula.
PS: affinché la ligasi espleti la sua funzione, è necessario che il nucleotide al 5' presenti un fosfato, non tre. |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 27 giugno 2011 : 14:47:28 Inserito il - 27 giugno 2011 : 14:47:28




|
Grazie Obarra! Quindi i nick che rimangono seppur riducendo il rendimento della procedura non la impediscono, tutto chiaro. Nel caso che hai disegnato tu quindi si tratta solo il vettore con la PA.
ps. considerando il 5' e il 3' nel tuo disegno, le posizioni dei "Fosfati presenti" non dovrebbero essere invertite? Al 5' intendo..perchè dal disegno sembrano siano localizzati al 3'..ma so bene che fare schemini con paint non è una passaggiata XD |
 |
|
|
0barra1
Utente Senior
   

Città: Paris, VIIème arrondissement

3847 Messaggi |
 Inserito il - 27 giugno 2011 : 14:54:45 Inserito il - 27 giugno 2011 : 14:54:45




|
Ahahha, dannazione, è vero, li ho disegnati invertiti, ovviamente si tratta di un errore, dal momento che i fosfati si trovano al 5' e su questo nessuno ha dubbi.
L'immagine corretta è questa:
Immagine:
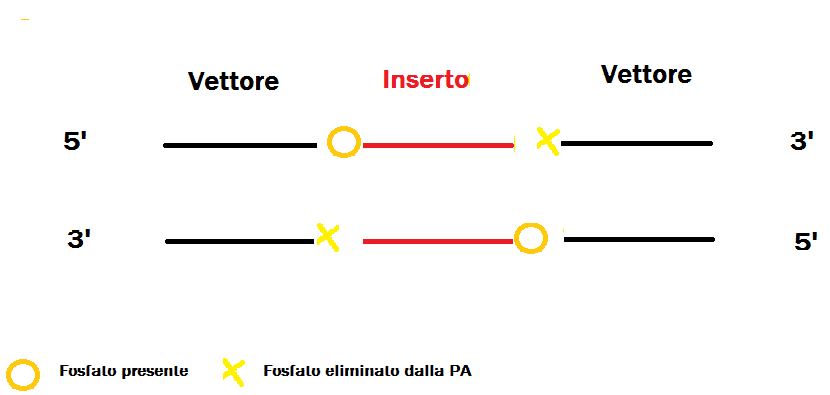
15,03 KB
Per evitare di indurre in errore gli utenti e non che in futuro consulteranno la pagina, ho eliminato dai miei file hostati su MoLab il disegno precedente. Ora dovrebbe essere tutto chiaro e coerente. |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 27 giugno 2011 : 15:01:26 Inserito il - 27 giugno 2011 : 15:01:26




|
Sì, tutto chiaro e coerente  |
 |
|
|
moni89
Nuovo Arrivato


60 Messaggi |
 Inserito il - 27 giugno 2011 : 16:42:19 Inserito il - 27 giugno 2011 : 16:42:19




|
Vi ringrazio per l'aiuto!   |
 |
|
| |
Discussione |
|







