| Autore |
Discussione |
|
|
rasta
Nuovo Arrivato

24 Messaggi |
 Inserito il - 15 febbraio 2008 : 20:10:07 Inserito il - 15 febbraio 2008 : 20:10:07




|
Ciao!!!!
Qualcuno saprebbe spiegarmi la SSH (Suppression Subtractive Hybridization)???!!!!
perchè il mio materiale è alquanto contrastante!!!!!!
grazie....  
|
|
|
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
rasta
Nuovo Arrivato

24 Messaggi |
 Inserito il - 16 febbraio 2008 : 11:08:40 Inserito il - 16 febbraio 2008 : 11:08:40




|
Grazie mille....ma io e l'inglese, PURTROPPO, non ci intendiamo.... e questo è un vero problema.....       |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 17 febbraio 2008 : 02:36:57 Inserito il - 17 febbraio 2008 : 02:36:57




|
Ok, vedo di spiegartelo io, però ti consiglio vivamente di provare almeno a leggerlo, anche se ci capisci 1/10 di quello che c'è scritto è sempre un buon esercizio ed è qualcosa con cui ti dovrai scontrare prima o poi (leggere o scrivere articoli in inglese).
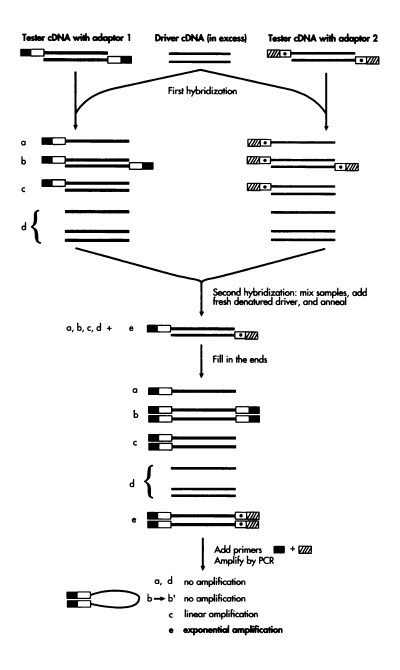
La figura più o meno parla da sola...
In pratica parti da 2 popolazioni di cDNA chiamate tester e driver (in eccesso).
L'idea è di ibridare la popolazione tester con quella driver e poi di separare il DNA ibridato da quello non ibridato. In questo modo hai "sottratto" i cDNA di tester che non sono presenti in driver (quello che non si è ibridato) e puoi vedere quali cDNA sono espressi in modo diverso nelle due popolazioni (es. prese da due tipi cellulari diversi)
Dividi in 2 la tua popolazione tester e ci attacchi degli adaptor diversi. Poi aggiungi un eccesso di driver e lo fai ibridare con i tuoi campioni dopo averli denaturati.
In pratica come vedi dal disegno avrai alcune molecole che non si ibridano (a), alcune che si ibridano con il tester (c) ed alcune che si ibridano tra di loro (b) e del driver in eccesso (d).
La popolazione a sarà arricchita in cDNA espressi differenzialmente.
a questo punto fai una seconda ibridazione, mischiando il risultato della prima ibridazione da entrambi i campioni con dell'altro driver denaturato.
Questa seconda ibridazione ti porterà ad avere a,b,c e d come sopra ed in più alcuni dei cDNA che erano in a (nei campioni 1 e 2) che si ibridano fra loro (e).
A questo punto completi i gaps alle estremità (funzionerà solo su b,c ed e) e ottieni un mix di a,b,c,d ed e.
Nota che nella figura i casi b e c sono mostrati solo con l'adapter 1 ma potrebbero anche essere con l'adapter 2.
Aggiungi primer per i 2 adaptor e, a parte possibile amplificazione lineare del caso c, avrai solo amplificazione esponenziale di e, che deriva dai tuoi cDNA espressi differenzialmente! |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
rasta
Nuovo Arrivato

24 Messaggi |
 Inserito il - 17 febbraio 2008 : 16:28:35 Inserito il - 17 febbraio 2008 : 16:28:35




|
Grazie mille chick80... non riuscivo a capire perchè, come mi hai poi sottolineato, i casi b e c fossero solo con l'adapter 1 e non col 2.
Per l'inglese ciò ho già sbattuto tantissime volte...specialmente alla specialistica!!!!
Volevo chiederti una cosa, anche se non è il luogo ideale... come sono le condizioni dei biologi molecolari in Australia e lì in Nuova Zelanda? perchè ho sempre sognato di venire a vivere lì..o almeno farmi qualche stage...
  
cmq grazie ancora.... |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 17 febbraio 2008 : 21:58:22 Inserito il - 17 febbraio 2008 : 21:58:22




|
| No problem. Le condizioni qui non sono niente male. Personalmente se vuoi starci per lungo tempo forse l'Australia è meglio (almeno per il clima e la densità di popolazione un pochino più alta, almeno sulla costa!) |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
rasta
Nuovo Arrivato

24 Messaggi |
 Inserito il - 17 febbraio 2008 : 22:05:50 Inserito il - 17 febbraio 2008 : 22:05:50




|
allora inizio a pensare seriamente di trovare un posto dove fare il dottorato....grazie mille!!!   |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
| |
Discussione |
|










