| Autore |
Discussione |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 15 aprile 2008 : 10:39:12 Inserito il - 15 aprile 2008 : 10:39:12




|
ciao a tutti,
in questi giorni mi trovo a dover rifare delle rappresentazioni che ho trovato in un articolo in bianco e nero.
La proteina è la CUZNSOD, cioè la superossido dismutasi, ed è un dimero.
Come posso fare per visualizzare solo uno dei due monomeri?
grazie
|
|
|
|
|
sverza
Nuovo Arrivato
Città: Milano

2 Messaggi |
 Inserito il - 17 aprile 2008 : 09:41:27 Inserito il - 17 aprile 2008 : 09:41:27





|
| Ciao! Esempio (ho scaricato da pdb.org 1AZV, dimero sod): in basso a destra del Pymol Viewer clicca su Selecting fino a quando non compare la scritta Chains. Quindi ti basta cliccare su di un qualsiasi residuo della catena che ti interessa ed essa interamente verrà selezionata. Ora puoi devisualizzare tale catena oppure tasto dx -> actions -> invert e ti seleziona tutto il resto, così da poterlo devisualizzare! |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 17 aprile 2008 : 19:41:28 Inserito il - 17 aprile 2008 : 19:41:28




|
grazie a tutti per i consigli...
ho risolto, alla prossima
ciao |
 |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 18 aprile 2008 : 11:21:55 Inserito il - 18 aprile 2008 : 11:21:55




|
altro dubbio:
usando pymol, come si fa a rendere piu visibili i label dei residui, cioè come faccio a fare apparire la scritta piu' distante dall'aa a cui si riferisce? questa cosa non l'ho trovata nella wiki di pymol.
grazie
|
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
 Inserito il - 18 aprile 2008 : 11:45:15 Inserito il - 18 aprile 2008 : 11:45:15





|

Se vai su Setting/Edit All... ti compare una finestra in cui puoi modificare manualmente tutti i parametri di visualizzazione di PyMol (parametri richiamabili pure con il comando set)
|
http://www.linkedin.com/in/dariocorrada |
 |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 18 aprile 2008 : 14:06:22 Inserito il - 18 aprile 2008 : 14:06:22




|
grazie, quindi secondo te come è meglio modificare il parametro relativo in modo da avere il nome dei residui piu visibile o comunque non coperto dai foglietti beta?
ciao
Immagine:
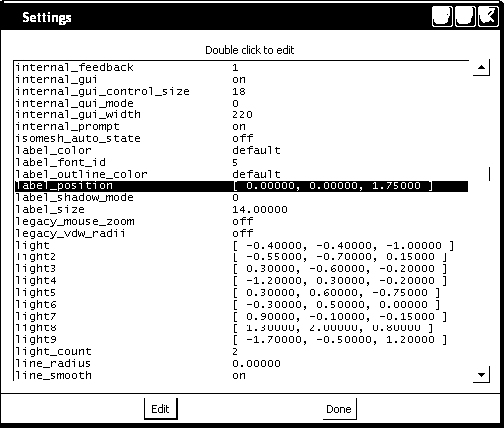
28,25 KB |
 |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 18 aprile 2008 : 14:09:38 Inserito il - 18 aprile 2008 : 14:09:38




|
mi accorgo solo ora che mi hai già indicato i parametri nell'immagine, sorry!
grazie
|
 |
|
| |
Discussione |
|






