| Autore |
Discussione |
|
|
tyler
Nuovo Arrivato

15 Messaggi |
 Inserito il - 16 aprile 2008 : 17:18:27 Inserito il - 16 aprile 2008 : 17:18:27




|
Ciao a tutti...ho un (tanto per cambiare) disperato bisogno di un consiglio...qualcuno di voi conosce/usa dei software che conteggino le cellule all'interno di immagini di istologia? (a.e. sezione colorata per PCNA, immagine scattata col PC (tiff, jpg...) un soft che conteggi le cellule totali e quelle PCNA positive? questo sarebbe il top...magari con la possibilità di cambiare le condizioni di conta in modo da riconoscere diversi marcatori...)
Spero di si ma temo sia solo un miraggio :(
Grazie a tutti!!
|
|
|
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 17 aprile 2008 : 01:55:57 Inserito il - 17 aprile 2008 : 01:55:57




|
Premesso che esistono diversi software commerciali e opensource che lo fanno, ricorda innanzitutto che la bontà dei risultati dipende essenzialmente dalla qualità dello staining.
Ti faccio un esempio di come farlo con ImageJ (software free e opensource)
1) File->Open e scegli il tuo file
Ho preso un'immagine a caso su Google di uno staining per c-fos.
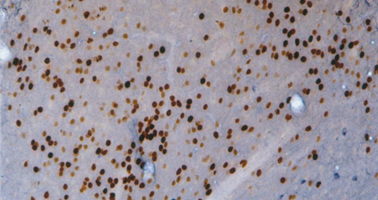
2) Scegli Process->Binary->Make binary e ottieni questo

3) Scegli Analyze->Analyze Particles. Puoi giocare un po' con i vari parametri. Assicurati di scegliere: "Display results" (che altrimenti è inutile!), e magari "outlines" nel campo show, che ti fa vedere cosa ha considerato come cellula e "summarize" che ti dà un riassunto dei risultati.
Ecco il risultato del nostro esempio con 329 cellule!
Nota che non ho ottimizzato i parametri. Ovviamente poi se parti da un'immagine più bella, magari la pasticci un po' prima del passaggio 2 etc, puoi migliorare i risultati (ad esempio quegli agglomerati di cellule vengono considerati come una cosa sola...).

Nota: se make binary non funziona bene puoi usare Image->adjust->threshold, ma assicurati che l'immagine sia a 8bit (Image->type->8bit)
   |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
tyler
Nuovo Arrivato

15 Messaggi |
 Inserito il - 17 aprile 2008 : 09:46:14 Inserito il - 17 aprile 2008 : 09:46:14




|
Grazie!!! appena ho 10 minuti liberi me li studio per bene...imageJ me lo avevano nominato già...devo assolutamente darci un'occhiata...magari farò qlk prova con delle immagini dove ho già contato, per vedere le discrepanze :)
Grazie ancora!!! |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
|
|
nyo84
Utente Junior


Prov.: Brescia
Città: Piancogno

239 Messaggi |
 Inserito il - 24 aprile 2008 : 18:11:07 Inserito il - 24 aprile 2008 : 18:11:07




|
| ma se io facessi una foto mentre conto con la camera di burker,posso utilizzare il programma lo stesso?? |
Fai attenzione quando leggi libri di medicina..potresti morire per un
errore di stampa... |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 24 aprile 2008 : 23:09:29 Inserito il - 24 aprile 2008 : 23:09:29




|
| Direi di sì, anche se alla fine mentre fai la foto, la scarichi su un computer (che probabilmente non sarà in camera sterile), e fai il tutto... probabilmente hai già contato le cellule a mano! :) |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Città: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 25 aprile 2008 : 12:48:02 Inserito il - 25 aprile 2008 : 12:48:02





|
Sì, ImageJ di NCBI é un buon software.
Stanno venendo fuori delle tecnologie molto interessanti collegate all'analisi di immagini prese al microscopio.
Se vi interessa l'argomento vi consiglio di leggere qualche review di Pepperkok, per esempio questa. |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
| |
Discussione |
|








