| Autore |
Discussione |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 11 maggio 2008 : 16:50:51 Inserito il - 11 maggio 2008 : 16:50:51




|
ciao a tutti,
come sa chi usa Pymol, questo programma offre la possibilitā di costruire molecole o peptidi.
Una volta messi in sequeza ad esempio 20 peptidi, che so che in una proteina formano una'alfa elica ad esempio, come faccio a fargli formare la struttura secondaria? Ho cercato anche nella wiki di pymol ma non ho trovato granchč....
grazie.
|
|
|
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Cittā: Monza

1303 Messaggi |
 Inserito il - 15 maggio 2008 : 11:25:37 Inserito il - 15 maggio 2008 : 11:25:37





|
Immagine:

168,78 KB
Prima di costruire il polipeptide dovresti settare il tipo di struttura secondaria, indicato dal tasto sotto l'elenco dei singoli residui.
In ogni caso, una volta finito, non dimenticare di farci una minimizzazione sulla struttura costruita
|
http://www.linkedin.com/in/dariocorrada |
 |
|
|
Bely
Nuovo Arrivato

38 Messaggi |
 Inserito il - 15 maggio 2008 : 16:11:55 Inserito il - 15 maggio 2008 : 16:11:55




|
Ma tu usi l'ultima versione? perche' io (che sono ahime' sempre con la vecchia) ho notato un "bug"... l'ho usato per costruire un pezzettino di elica, ma mi mette la lunghezza del legame peptidico a 1.47 
Per questa funzione c'e' anche il server PepBuild (http://www.imtech.res.in/bvs/pepbuild/), pero' in questo momento dev'essere un po' intasato, perche' non riesco ad usarlo  |
 |
|
|
RNA world
Utente Junior



370 Messaggi |
 Inserito il - 16 maggio 2008 : 13:20:45 Inserito il - 16 maggio 2008 : 13:20:45




|
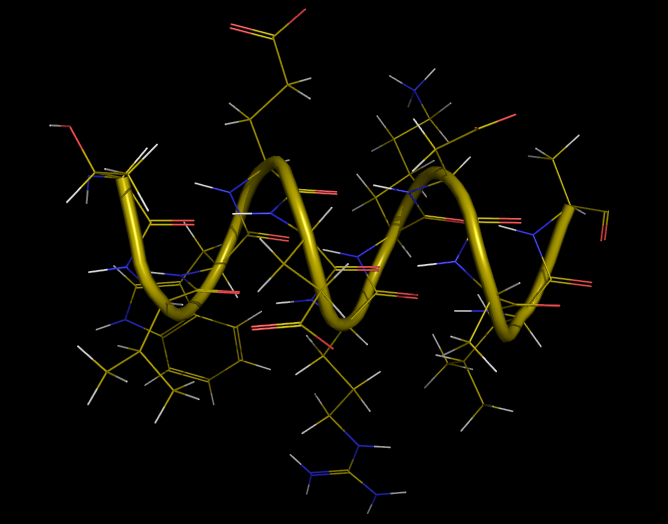
grazie ...
in effetti bastava solo selezionare la voce "alfa elica".
ciao
Immagine:
99,1 KB |
 |
|
| |
Discussione |
|








