Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
kondalord
Nuovo Arrivato

Prov.: Bari
Città: altamura

68 Messaggi |
 Inserito il - 12 settembre 2008 : 22:14:00 Inserito il - 12 settembre 2008 : 22:14:00





|
non ho capito una cosa sulle genoteche, una volta create come si utilizzano?
cioè se mi serve un particolare gene, per trovarne il clone devo comunque piastrare su un terreno selettivo o qualcosa che me lo identifichi giusto?
|
|
|
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 13 settembre 2008 : 17:19:51 Inserito il - 13 settembre 2008 : 17:19:51





|
Diciamo che una genoteca non si fa da un giorno all altro in laboratorio .... in genere si comprano e si conservano nel -80°C gli stock in glicerolo nal caso siano stati usati vettori BAC trasformati in E.Coli.
Queste genoteche sono delle gliglie che contengono plasmidi dove ciascun plasmide ha un suo nome e cognome (tipo le caselle della battaglia navale),ovviamente quando si fa una cosa del genere dev essere un qualcosa che sia standardizzata, nel senso che se io ho usato un BAC ( questo vale anche per le genoteche fagiche,ma prendero' come riferimento un vettore BAC ),con un certo nome che ho preso in quella determinata posizione,anche chi ha la genoteca in australia o ovunque sia,una volta avute le cordinate del punto in cui si trova se lo potra' cercare nella sua genoteca.
Ora chi ha la fortuna di avere queste genoteche,non c'e' bisogno di piastrare....utilizzare sonde etc....perche' sarebbe ogni volta un lavoro abnorme per avere una sequenza ,che poi in teoria con la PCR potresti avere quello che vuoi ,quindi per evitare questo esistono delle banche dati in cui sono stati inseriti i nomi dei BAC e tutto cio' che la sequenza contiene,ovviamente non in tutti perche' come tu ben sai il genoma umano sebbene e' stato ampiamente sequenziato e mappato presenta ancora dei grossi buchi; ,quindi per evitare questo esistono delle banche dati in cui sono stati inseriti i nomi dei BAC e tutto cio' che la sequenza contiene,ovviamente non in tutti perche' come tu ben sai il genoma umano sebbene e' stato ampiamente sequenziato e mappato presenta ancora dei grossi buchi;
tra queste banche dati c'e' la UCSC genome browser che permette di ricavare il BAC d interesse
Ora ti mostro i passaggi
qui mettiamo il nome del nostro gene che dobbiamo cercare nella library
Immagine:
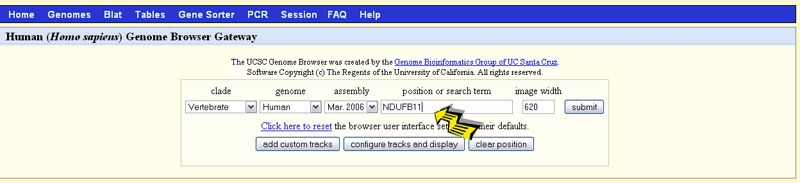
52,71 KB
Immagine:

118,66 KB
a questo punto ci dara' gia' delle informazioni come ad esempio su quale chromosoma mappa il tipo di proteina etc...
selezioniamo il nostro gene che ci porta ad un altra schermata
Immagine:
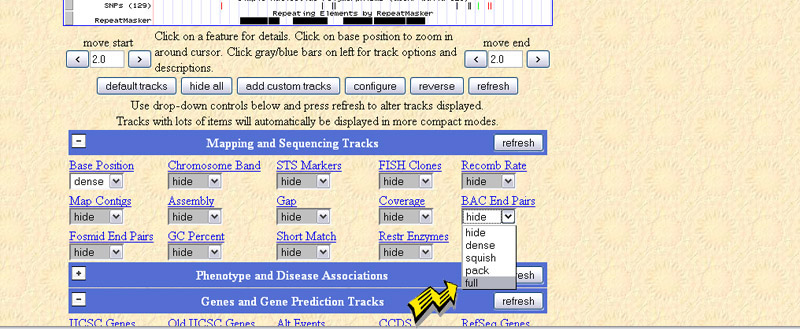
135,53 KB
qui sotto ci sono diverse opzioni scegliere,ma a noi cio' che serve e' che mi visualizzi il nome del clone che porti il mio gene ,quindi vado su BAC end pairs e clicco Full ,a questo punto diamo un refresh e ci apparira' il nome del nostro BAC che contiene il gene d interesse
Immagine:
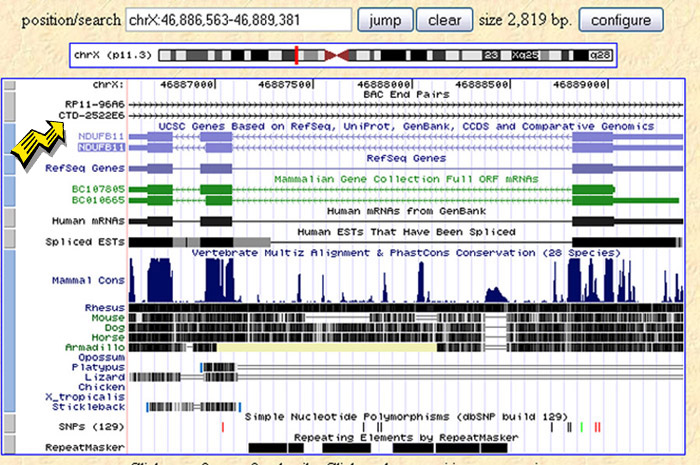
162,29 KB
qui ne mostra 2 perche' le genoteche umane sin ora fatte con i BAC sono di 2 tipi quella della RP e CTD ( non mi chiedere nel dettaglio  ) ,quindi avremo che il nostro BAC si chiama RP11-96A6,scegliamo la piastra e il pozzetto con questa serie...e a questo punto basta piastrare far crescere i batteri and the play is made ) ,quindi avremo che il nostro BAC si chiama RP11-96A6,scegliamo la piastra e il pozzetto con questa serie...e a questo punto basta piastrare far crescere i batteri and the play is made
 |

|
 |
|
|
kondalord
Nuovo Arrivato

Prov.: Bari
Città: altamura

68 Messaggi |
 Inserito il - 13 settembre 2008 : 22:04:01 Inserito il - 13 settembre 2008 : 22:04:01





|
AVE CESARE!
grazie!!! |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|










