Come funziona l’ingegneria genetica? Spaccando tutto… per rimettere tutto a posto
Non sono un appassionato di CSI: ciò nondimeno, rimango sempre affascinato durante la puntata occasionale che guardo. Coloratissime soluzioni vengono infilate dentro delle provette, a loro volta utilizzate scenograficamente in incredibili macchine (molto spesso, comuni apparecchiature da laboratorio che non c’entrano NULLA con quanto viene detto), per restuire in tempi record incredibili quantità di dati, visualizzati con interfacce che alla Pixar costerebbero giorni di rendering.
La fantascienza forense oltrepassa di molto le capacità di un laboratorio di analisi che esiste davvero: ma la biologia molecolare batte trecento a zero anche i più bravi sceneggiatori. In laboratorio possiamo fare cose che non si sognano nemmeno: oggi ve ne voglio raccontare una.
La biologia molecolare è – nè più nè meno – l’arte dell’autofficina trasportata nei sistemi biologici. E’ un insieme di tecniche e metodiche che permettono al biologo molecolare di utilizzare composti chimici e piccole macchine biologiche (enzimi) per interrogare i sistemi biologici o per modificarli, o per costruire dei programmi (genetici) che possano essere poi interpretati dai sistemi biologici stessi. Insomma, il biologo molecolare è il perfetto incrocio tra un programmatore e un carrozziere, i cui attrezzi sono semplicemente qualche centinaio di milioni di volte più piccoli.
Sono rimasto molto colpito da un articolo apparso ad agosto su Nature Methods, che è una specie di Pimp my ride cartaceo per noi biotecnologi. Un tema caldo è come intervenire per modificare i genomi degli organismi complessi in modo mirato, senza spaccare tutto a casaccio (l’approccio che andava per la maggiore): lo utilizzerò per trascinarvi nel mio mondo e darvi un’infarinatura di come effettivamente vengano fatte tutte le cose dai nomi altisonanti che sciorinano alla televisione.
Come dei bravi alunni che non copiano a scuola, gli organismi superiori hanno perso la capacità di effettuare la ricombinazione omologa di pezzi di DNA introdotti articiosamente dall’esterno con il loro DNA genomico. Cioè, se con la mia DNA-officina miniaturizzata costruisco un pezzetto di DNA (contenente un bellissimo programma genetico tutto nuovo), posso sì infilarlo in un organismo – ma questo andrà a mettersi (nel genoma) dove più gli pare e piace (dove probabilmente non funzionerà come deve, come insegna Murphy). Alcuni organismi (come Saccharomyces cerevisiae) sono invece specializzati nel sostituire col nuovo codice il loro vecchio codice, a patto che sia contenuto tra pezzi di codice che non deve cambiare (l’omologia delle sequenze esterne serve ad indirizzare correttamente il frammento di DNA affinchè raggiunga la posizione voluta). Sostituendo l’intero pezzetto di DNA, la modifica che era contenuta nella porzione centrale (fatta da noi) andrà a sostituire quella presente in origine. E’ possibile fare una cosa del genere anche in organismi più semplici dei lieviti come i topi (e da quest’anno, anche nei ratti): questa sciocchezza ha consegnato il Nobel ad un nostro connazionale, Capecchi.
Una volta operata la modifica, è possibile selezionare le cellule in cui questa è avvenuta in modo corretto, propagarle, ed utilizzarle per continuare a lavorare in provetta (in vitro), oppure utilizzarle per generare degli esseri viventi (in vivo) che abbiano nel loro patrimonio genetico la modifica da noi introdotta.

Utilizzare le cellule staminali embrionali (ES cells) sfruttando la loro capacità di effettuare ricombinazione omologa (paragonabile alle abilità in matematica di Pierino) era una tecnica consolidata solo nel topo, fino all'anno scorso. Nella foto, papà ratto (destra) è una chimera: un organismo costituito da cellule con un genoma diverso (riconoscibili facilmente dal diverso colore del pelo). Chimera costruita in laboratorio, iniettando le cellule modificate dai ricercatori dentro un embrione di ratto. fonte. Nature
E’ imperativo però sviluppare degli attrezzi che ci permettano di intervenire nei sistemi che sono refrattari a questo tipo di intervento, per operare modifiche mirate nel genoma degli organismi al fine di produrre modelli da utilizzare in ricerca, ma non solo. Molto recentemente è stata presentata la tecnologia delle Zinc Finger Nucleases, che permette di operare in modo preciso nel genoma: un sogno fino a poco tempo fa. Le ZFN sono proteine costruite a tavolino affinchè possiedano due tipi di attività: riconoscimento di una specifica sequenza di DNA (la sequenza che vogliamo modificare!) e taglio del DNA una volta avvenuto il legame.
Pezzettini molto corti di sequenza possano essere letti da proteine che, scorrendo sull’elica del DNA come un dito su una superficie con dei rilievi, possano riconoscere “cosa c’è scritto”, legandosi saldamente ad alcune sequenze piuttosto che ad altre. Tra i motivi proteici in grado di leggere il DNA, un tipo ha proprio la forma di un “dito” che scorre lungo l’elica, e si chiama per l’appunto zinc finger, ossia “dito di zinco” (le “dita” sono mantenute in struttura da ioni zinco).
Montando ad hoc un dito dopo l’altro, si possono costruire delle proteine artificiali con una specificità unica rispetto ad una sequenza, tanto complessa tante dita utilizzo per il suo riconoscimento. Ma non è finita qua: unendo queste ad una proteina che di mestiere spacca il DNA (e volendo anche mettere la ciliegina sulla torta, facendo pure in modo che il tutto non funzioni a meno che dall’altra parte dell’elica non si sia legata anche una ZFN “sorella”) si apre un bel buco nel DNA.
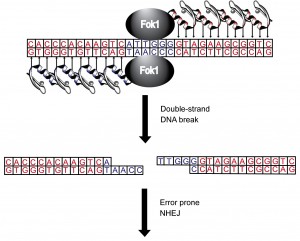
Questo più o meno è quello che viene descritto dal desto. FokI è il nome dell'endonucleasi (un enzima che taglia all'interno - endo - dell'acido nucleico), e funziona solo come dimero. Un po' come un paio di forbici: tagliare un filo con una sola forbice (?) è arduo, mentre se le forbici sono montate il gioco è fatto. La parte di zinc finger dice a FokI DOVE andare a tagliare. Fonte: twiw.tv
Ma non eravamo partiti con un’idea diversa.. tipo modificare il DNA? Per ora abbiamo solo spaccato tutto.. dove vogliamo arrivare? Beh – quando vi tagliate, che cosa succede? La ferita si chiude, e guarisce. Per il DNA è la stessa cosa: le ZFN servono a sfruttare i naturali meccanismi di riparazione del DNA, ed utilizzarli per “riparare il DANNO” utilizzando come templato il DNA che abbiamo prodotto nella nostra piccola DNA-autofficina-laboratorio di biologia molecolare.
La ferita nel DNA indotta dall’azione delle ZFN viene riparata in due modi differenti a seconda dell’immediata disponibilità – o meno – di un templato da cui copiare. Nel primo caso, la riparazione è priva di errore (o meglio, vengono incorporate nel DNA le modifiche presenti e introdotte da noi). Nel secondo, gli enzimi che effettuano il lavoro non hanno altra possibilita che “reincollare il tutto alla meno peggio”.
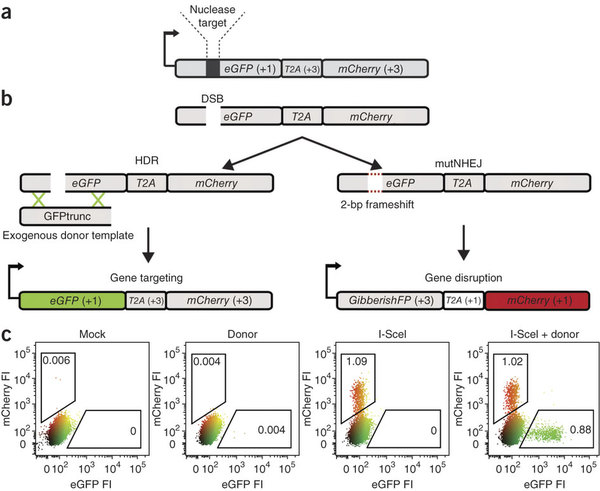
Le sequenze di DNA destinate a dar vita a proteine vengono trascritte in RNA e "lette" in triplette: tre lettere per un amminoacido. La cattiva nucleasi (l'enzima che "buca" il DNA) rompe la sequenza codificante di eGFP (una proteina fluorescente verde). A noi il compito di ripararla, per vedere cosa succede. Se la riparazione avviene per ispirazione dal DNA che abbiamo fornito noi dall'esterno, il gene viene riparato e la cellula sarà verde. Altrimenti, gli enzimi riparativi introdurranno una variazione della cornice di lettura, che porterà alla traduzione di mCherry, un'altro gene reporter il cui superpotere è quello di essere.. rosso. Ma quante ne sanno!
Avere ottenuto questo risultato è un passo gigante, ma studiare un sistema certosino per scoprire in che proporzione avvengano questi fenomeni, e fare in modo che basti la semplice osservazione di un colore è assurdamente geniale. L’utilizzo di questi geni reporter, che hanno la sola funzione di essere manifestamente evidenti, è naturalmente presa con ironia dagli organi di stampa (la ricordate quella del cane verde?), ma ci sono più cose che si possono fare con eGFP di quante non se ne immaginino nemmeno in filosofia. E in CSI.
Tag:CSI, DNA, Elettroporazione, genetica, genoma, GFP, Infezione, Ingegneria genetica, legge di Murphy, Lentivirus, Mario Capecchi, mCherry, Nature, Nature Methods, Nobel, Pimp my ride, proteine fluorescenti, Zinc finger nuclease
seguici anche su Facebook!



ndd (nota del demente)
Capecchi italiano… e’ scappato con la mamma dallo zio americano a 12 anni…
T2A? Che cos’e'? Una nuova tecnologia? L’hanno provata con dei vettori vuoti e correndo dei gel senza corrente come controllo?
[...] di formare cellule provenienti da tutti e tre i foglietti embrionali. Erano quelle che utilizzavamo qualche tempo fa per produrre topi geneticamente modificati, insomma. Pro: ci possiamo fare un sacco di cose. [...]
[...] dell’ordine interno sarebbe meglio farlo all’interno di noi stessi medesimi…) dalle cellule staminali, anche se è molto meno costoso ottenere questi elementi da delle fabbriche naturali di queste [...]
[...] [...]
Segnalo un refuso: “anche in organismi più semplici dei lieviti come i topi” dove immagino volessi scrivere “complessi”.