Una giornata in laboratorio: la folle corsa del DNA
Nella nostra ultima visita in laboratorio, abbiamo estratto DNA da un pezzo di tessuto. Oggi invece questo DNA lo utilizzeremo praticamente per effettuare degli esperimenti! Siete pronti?
Uuuh! E che sarà mai?
Le cose che ci può raccontare il DNA sono tantissime: non posso esaurirle in un post e nemmeno in un libro. Stavolta ci faremo raccontare dal DNA qualcosa sul suo contenuto utilizzando una tecnica chiamata PCR (reazione a catena della polimerasi). Questo però non è un post sulla PCR (sulla quale potete documentarvi su Molecularlab). In sintesi, vi basti sapere che
- La PCR sfrutta la complementarietà dei filamenti di DNA per ottenere una amplificazione mirata di alcuni pezzettini di DNA, utilizzando l’enzima che copia il DNA (la DNA Polimerasi)
- E’ capace di produrre miliardi di molecole di amplificato partendo da una sola copia di originale
- E’ stata inventata da un pazzo/genio assoluto di nome Kary Mullis, premio Nobel per la chimica 1993. Un personaggio fantastico di cui vi consiglio assolutamente la lettura della spassosissima autobiografia, Ballando nudi sul campo della mente (Dancing naked on the mindfield)
Quest’oggi facciamo una PCR per scoprire se nel nostro DNA è presente (o no) una mutazione: sapendo a priori cosa ci attendiamo, è possibile disegnare un esperimento che risponda al nostro interrogativo.
Prepariamoci a fare una PCR!
Eccomi pronto! Puntali, pipette, reagenti e provettine nelle quali effettuare la reazione. E naturalmente i conti per mischiare tutto quanto assieme (da quando uso un foglio Excel la mia vita è cambiata! ^_^).
Tutto può essere un templato per una reazione di PCR. Anche le mie mani!
Dopo aver assemblato la reazione, aggiungo al controllo negativo (una provetta nella quale non viene aggiunto DNA per verificare che non ci siano contaminanti nei reagenti) un controllo negativo aggiuntivo utilizzando quello che hanno toccato le mie mani: non deve avvenire nessuna amplificazione!
Un aspetto interessante della PCR è la sua estrema sensibilità. Il volume della mia reazione è tipicamente 15 microlitri (milionesimi di litro): una goccia molto piccola. Se io vuotassi questa goccia nel volume di acqua che occuperebbe una piscina olimpionica (1), mescolassi per bene e alla fine prelevassi un microlitro (un milionesimo di litro) da questo volume di acqua (circa due milioni e cinquecentomila litri), ci troverei dentro decine di copie di pezzettini di DNA. Voi potete ben immaginare che cosa significhi solo aprire una provetta dopo l’amplificazione in una stanza chiusa: gli amplificati vanno ovunque e naturalmente non possiamo permetterci che contaminino nè la reazione nè i nostri campioni!
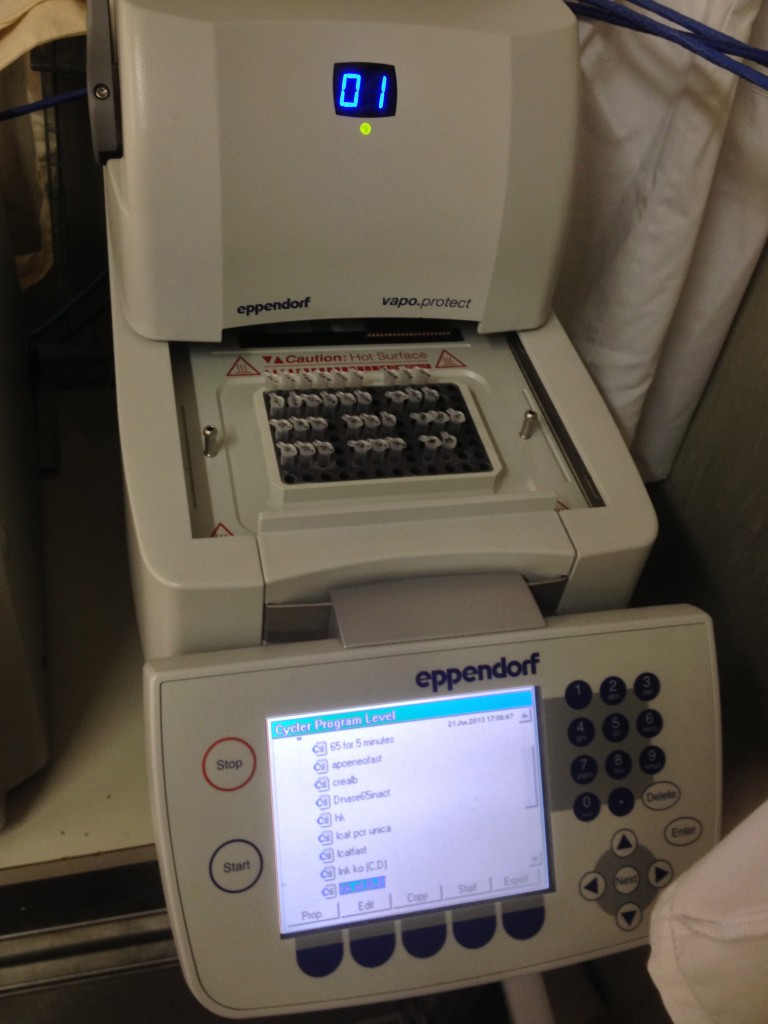
Una volta finito, metto le provette nel blocco di metallo del termociclatore: uno strumento che cambia la temperatura secondo uno schema impostabile, che utilizzo per far avvenire la reazione.
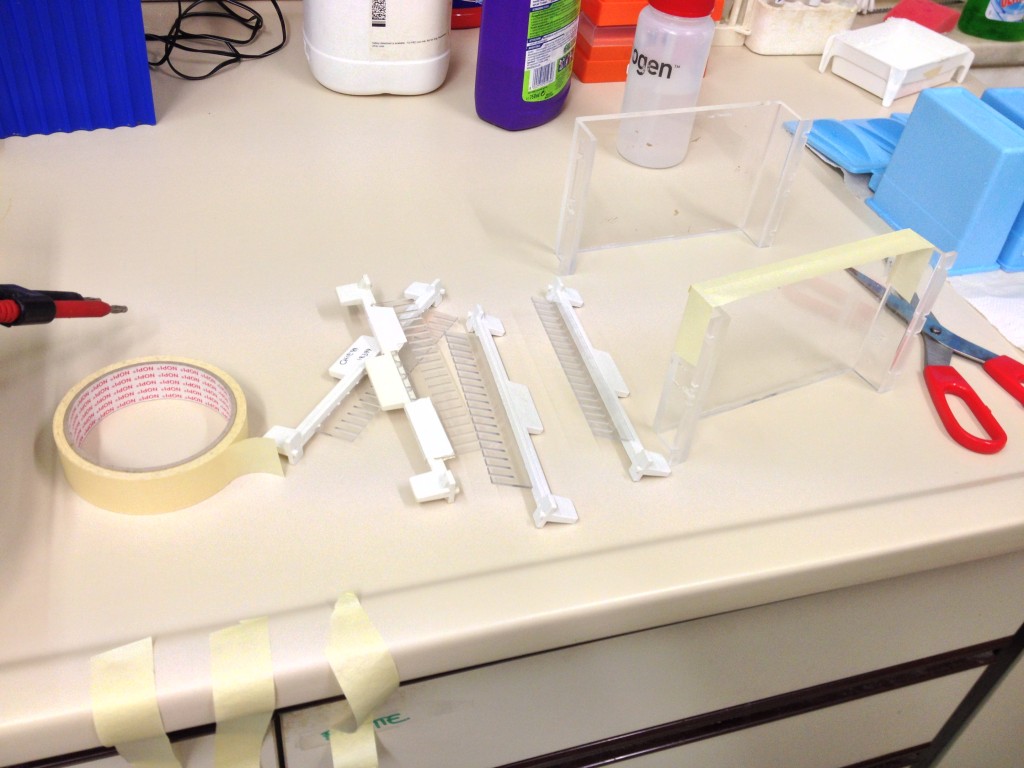
Tutto quanto serve per colare un gel di agarosio.
Mentre la reazione procede, assemblo tutto quello che mi serve per colare un gel di agarosio (fratello del gel di poliacrilamide che abbiamo già utilizzato per correre le proteine). Utilizziamo l’agarosio perchè una volta polimerizzato produce una rete molecolare più grande, più congeniale alla separazione del DNA per dimensione.

Ingredienti per un gel d'agarosio: agarosio.
I conti ci dicono che dobbiamo pesare due grammi e 52 di agarosio: stavolta siamo stati bravi!

Nella beuta (l’affare di vetro) l’agarosio sta vorticando insieme al TAE (2), il tampone in cui preparo il gel e serve anche da tampone di corsa. Il tutto aiutato da un’ancoretta magnetica!

Cotto a puntino!
Dopo la necessaria bollitura, questo è l’aspetto che deve avere il gel per essere colato!

Durante la bollitura, un po’ di acqua evapora necessariamente. Dato che facciamo le cose per bene e il gel lo vogliamo con la percentuale di agarosio/TAE che vogliamo noi, ripristino il volume di acqua che manca con acqua distillata. Tarare la beuta prima di bollire vi permette di rilassarvi e non preoccuparvi se lasciate il gel a bollire un po’ più del dovuto.
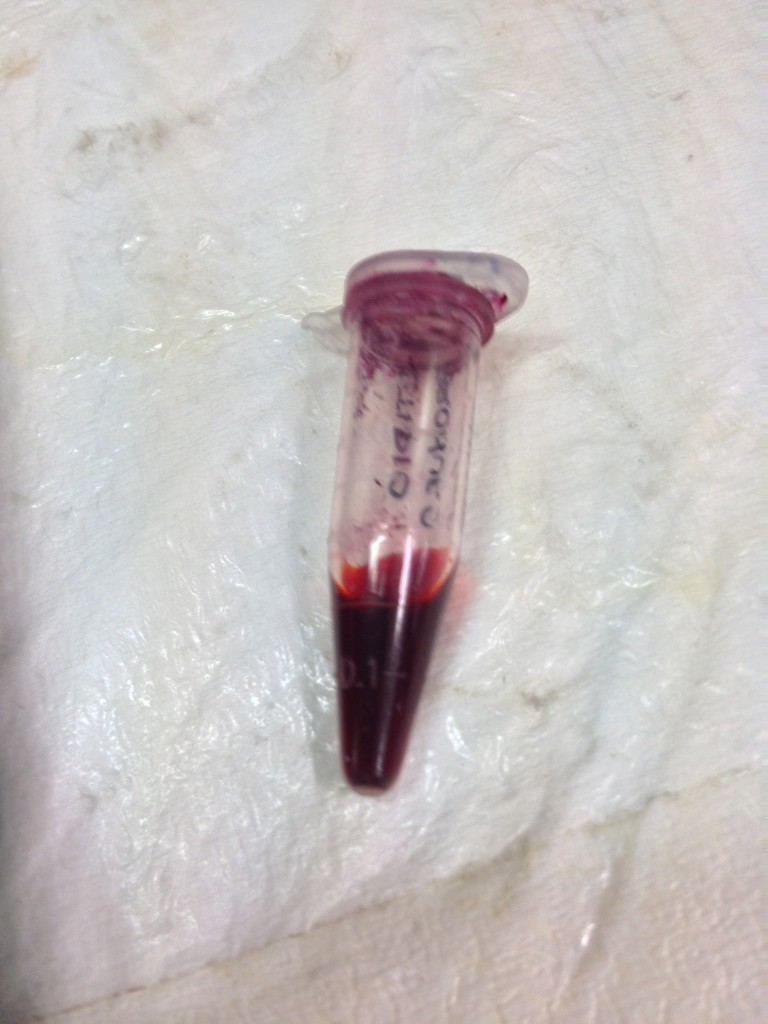
Lo spauracchio di grandi e piccini: etidio bromuro!
Prima di colare, aggiungiamo un composto chimico che servirà per la rilevazione del DNA una volta terminata la corsa. Rosso, famoso e cancerogeno: etidio bromuro!

Ho appena colato il gel. Tra una quindicina di minuti sarà polimerizzato.
Questo è l’aspetto del gel appena colato.

Ecco fatto! Pronti per caricare.
Questo è l’aspetto del gel una volta polimerizzato. Vedete che si è opacizzato? Il pettine che vedete verrà rimosso e lascerà spazio a dei pozzetti in cui verrà inserito il campione di DNA.
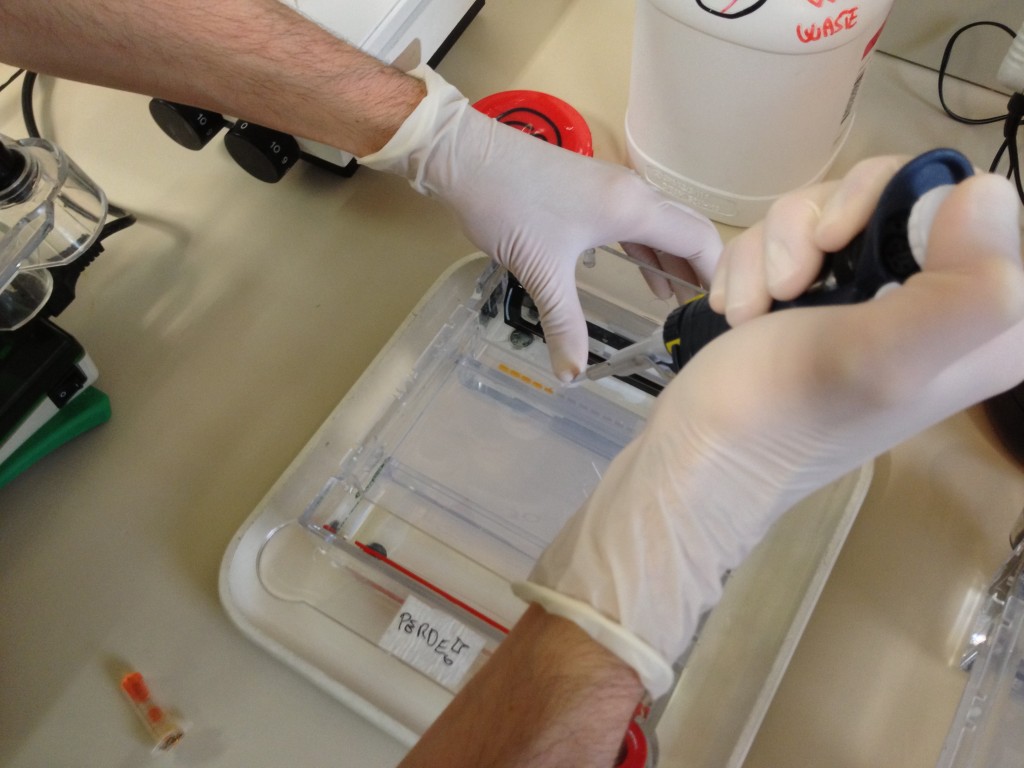
Carico nei pozzetti 10 microlitri di reazione di PCR (tenendone 5 per eventuali catastrofi) nel gel, utilizzando una soluzione più densa dell’acqua (glicerolo ^_^) che è anche colorata. Preferisco l’Orange G (il nome di questo colorante) perchè non si sovrappone a dove probabilmente c’è il DNA (come spesso fanno i coloranti blu) e perchè è arancione! *.*
Che cosa succede ora? Spinto dal campo elettrico (il DNA ha una carica elettrica negativa, per cui si muoverà verso il polo positivo), i pezzetti di DNA correranno tanto più velocemente tanto più saranno di piccole dimensioni. [Più informazioni su Molecularlab]

Questo è l’aspetto di un gel dopo la corsa. Il colorante ha “migrato” spinto dal campo elettrico: il DNA (che non possiamo ancora vedere) avrà fatto lo stesso.
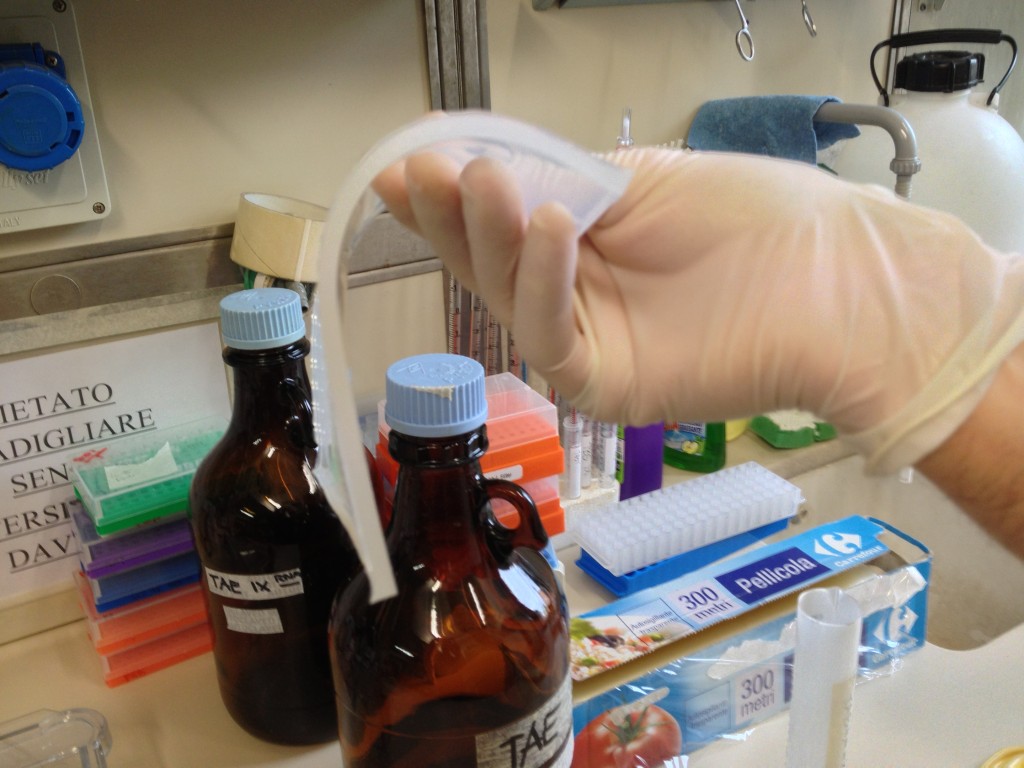
Una volta terminata la corsa, prendiamo il gel per vedere cosa è successo!
Ecco che cosa succede a fare troppo gli sciocchi! ._.

Finalmente, siamo pronti ad osservare il risultato delle nostre fatiche!
Questo strumento è un transilluminatore, dotato anche di videocamera. Il gel (che vedete) è posto sopra ad un affare pieno di lampade UV: le lampade eccitano il bromuro di etidio (il colorante rosso di prima) che si è legato al DNA nel gel, il quale riemette nel visibile: sarà proprio questo segnale a rivelare la presenza del DNA nel gel.

Foto del gel in luce visibile
Questa è l’immagine del gel illuminato dalla luce visibile: riuscite a vedere qualcosa?
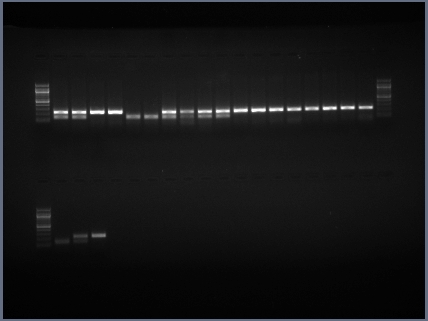
UV!
Questa invece è la fotografia dello stesso gel illuminato da luce UV. Naturalmente, gli UV non sono visibili (nemmeno dalla fotocamera che opera in luce visibile), ma l’etidio bromuro, eccitato dalla luce UV, riemette in visibile. E’ questo segnale che viene catturato dalla fotocamera: come potete vedere il gel non è affatto vuoto! Dall’alto verso il basso, i filamenti di DNA si sono disposti dal più lungo al più corto. Come potete vedere, nella stessa corsia possono convivere pezzi di DNA di diverse lunghezze, le bande semplicemente si “sistemano” ad altezze differenti. La disposizione delle bande che vedete e la loro interpretazione sono la risposta che stavamo cercando!
Piermatteo
(1) Le dimensioni della piscina, qui. Il conto naturalmente è spannometrico: assumendo una piscina olimpionica da 10 corsie di un volume di circa 2,5 milioni di litri, e assumendo una PCR con efficienza al 100% di cento molecole di partenza e 40 cicli di amplificazione, otterremmo circa 22 copie per microlitro (una volta diluita la PCR nella piscina).
(2) TAE: Tris, acido acetico, EDTA. Uso questo invece del TBE perchè lo posso preparare 50X (anzichè 10X): molto meno sbattimento! :)
Tag:agarosio, DNA, DNA polimerasi, elettroforesi, etidio bromuro, Kary Mullis, laboratorio, PCR, ricerca
seguici anche su Facebook!


Ma che bello questo post!
La prossima volta che mi imbatterò in un biologo che parla di PCR almeno saprò con cosa combina operativamente.
Trovo molto interessante questa specie di format da “ricetta illustrata” per cercare di far vedere ai non addetti ai lavori come funzioni il lavoro in laboratorio.
Sottoscrivo :)
Leggo con molto piacere questi post di “tecnica di laboratorio applicata”, sono… come dire… belli! Si, belli rende.
Uno scorcio su un lato della ricerca di cui non si sente parlare quasi mai, il “lavoro sporco” dietro le analisi e le pubblicazioni, lavoro che fà anche trasparire le competenze di chimica, fisica e perchè no, di meccanica necessarie a compiere questo lavoro.
Vero che ne pubblicherete altri di post così??? :)
Ciao!